Comprobar diferencias en la microbiota duodenal al diagnóstico de la enfermedad celíaca (EC) en relación con un grupo control.
Material y métodosSe obtuvieron muestras de biopsias duodenales en 11 pacientes con EC al diagnóstico y en 6 controles. Se analizó la microbiota duodenal total así como la perteneciente al género Lactobacillus mediante la técnica molecular PCR-electroforesis en gel con gradiente desnaturalizante (DGGE). Los patrones de bandas obtenidos en los geles resultantes fueron analizados para determinar las diferencias presentes entre la microbiota de pacientes con EC y de los controles (FPQuest 4.5), mientras que los índices ecológicos (riqueza, diversidad y habitabilidad) fueron calculados con el programa Past versión 2.17.
ResultadosLa microbiota intestinal de los individuos con histología Marsh 3c presentó similitud del 98% y fue diferente del resto de pacientes celiacos. Las principales diferencias se obtuvieron en los índices ecológicos pertenecientes al género Lactobacillus, con importante reducción de especies en los celiacos respecto al grupo control (riqueza, diversidad y habitabilidad). En los pacientes con EC las bandas principalmente fueron catalogadas con las especies Streptococcus, Bacteroides y E.coli. En los controles las bandas predominantes fueron Bifidobacterium, Acinetobacter y Lactobacillus; sin embargo, los Streptococcus y Bacteroides fueron más bajos.
ConclusionesLos índices ecológicos aplicados al género Lactobacillus fueron significativamente reducidos en los pacientes celíacos. Los casos con mayor afectación histológica presentaron una microbiota duodenal similar.
To establish whether the duodenal mucosa microbiota of children with active coeliac disease (CD) and healthy controls (HC) differ in composition and biodiversity.
Material and methodsSamples of duodenal biopsies in 11 CD patients were obtained at diagnosis, and in 6 HC who were investigated for functional intestinal disorders of non-CD origin. Total duodenal microbiota and the belonging to the genus Lactobacillus using PCR-denaturing gradient gel electrophoresis (DGGE) were analysed. The banding patterns obtained in the resulting gels were analysed to determine the differences between the microbiota of CD patients and HC (FPQuest 4.5) while environmental indexes (richness, diversity and habitability) were calculated with the Past version 2.17 program.
ResultsThe intestinal microbiota of patients with Marsh 3c lesion showed similarity of 98% and differs from other CD patients with other type of histologic lesion as Marsh3a, Marsh3b and Marsh2. The main differences were obtained in ecological indexes belonging to the genus Lactobacillus, with significant richness, diversity and habitability reduction in CD patients. In CD bands were categorized primarily with Streptococcus, Bacteroides and E.coli species. In HC the predominant bands were Bifidobacterium, Lactobacillus and Acinetobacter, though the Streptococcus and Bacteroides were lower.
ConclusionsThe celiac patients with major histological affectation presented a similar microbiota duodenal. The ecological indexes applied to the genus Lactobacillus were significantly reduced in CD.
La enfermedad celíaca (EC) es un trastorno inflamatorio del intestino delgado caracterizado por una intolerancia permanente al gluten de la dieta y que se manifiesta en individuos con predisposición genética1. La enfermedad puede desarrollarse a cualquier edad y con manifestaciones clínicas heterogéneas, aunque aparece típicamente en la infancia temprana. Los pacientes con EC pueden ser asintomáticos, presentar únicamente síntomas extraintestinales o padecer formas silentes de celiaquía. En la actualidad la EC con síntomas gastrointestinales clásicos se diagnostica objetivando la atrofia vellositaria, la hiperplasia de las criptas y la infiltración linfocitaria.
La EC es un trastorno multifactorial en el que interactúan factores genéticos y ambientales. Entre los factores ambientales se encuentran la exposición temprana al gluten, la lactancia materna de corta duración, las infecciones intestinales y las alteraciones de la microbiota.
Recientemente la literatura científica ha descrito alteraciones en la composición de la microbiota en pacientes celíacos2. Hasta el momento las diferencias en la composición de la microbiota y en metabolitos asociados entre pacientes celíacos y controles sanos (CS) han sido descritos en base a muestras fecales3,4. También se ha descrito una mayor diversidad bacteriana y diferencias en varios grupos de bacterias en la microbiota duodenal de pacientes pediátricos con EC5–7. Sin embargo, otros estudios no consiguieron detectar diferencias significativas entre la microbiota de niños celíacos y la de CS8–10. Cheng et al.10 describieron una composición similar de la microbiota en niños celíacos y CS, pero observaron diferencias significativas en un perfil subpoblacional de 8 grupos bacterianos a nivel de género que podría desempeñar un papel específico en el trastorno epitelial de la EC.
Se han descrito cambios en la microbiota duodenal de pacientes celíacos adultos y pediátricos tanto con enfermedad activa5,9,11 como cuando siguen una dieta sin gluten12. Wacklin et al.13 encontraron diferencias entre la composición y la diversidad de la microbiota intestinal de adultos con síntomas intestinales clásicos de EC y las de adultos con síntomas extraintestinales, indicando que la composición de la microbiota residente en la mucosa duodenal de los pacientes difería basándose en las manifestaciones de la EC.
El objeto de nuestro estudio era establecer si la microbiota duodenal de los niños con EC activa y la de los controles difieren en su composición y biodiversidad para explicar las diferencias entre la microbiota de los pacientes pediátricos celíacos y la de los controles sanos.
Materiales y métodosSujetosSe recogieron biopsias duodenales al diagnóstico de niños con EC que seguían una dieta normal con gluten, y de niños sanos. El grupo de niños sanos lo constituían pacientes que estaban siendo evaluados por trastornos funcionales gastrointetsinales no relacionados con la enfermedad celíaca ni el gluten. Todos los sujetos sanos tenían marcadores serológicos de EC negativos y una mucosa normal en la biopsias del intestino delgado (Marsh0)14. Los pacientes con EC tenían tanto marcadores serológicos positivos (anticuerpos antitransglutaminasa tisular IgA, anticuerpos antigliadina deaminada IgG y anticuerpos antiendomisio IgA) como lesiones histológicas en la biopsia duodenal.
El estudio se llevó a cabo conforme a los principios de la Declaración de Helsinki y siguiendo los EEC Good Clinical Practice Guidelines (Guía de Buena Práctica Clínica, documento 111/3976/88, julio de 1990), y dentro de marco legal vigente en España para regular la investigación clínica en seres humanos (Real Decreto 561/1993). El protocolo del estudio fue aprobado por el Comité de Ética del CSIC y por el hospital.
Se obtuvo el consentimiento informado de los padres de los niños incluidos en el estudio. Ningún caso incluido en el estudio había recibido tratamiento antibiótico en los 2 meses previos a la endoscopia. Se tomaron 4 biopsias de la mucosa duodenal de cada paciente. Dos muestras de cada paciente se enviaron para examen histológico empleando la clasificación de Marsh, y otras 2 se congelaron de inmediato tras su obtención y se almacenaron a –20°C hasta su procesamiento para estudio bacteriológico. Estas últimas se suspendieron en 1ml de PBS y se centrifugaron a 1.000g durante 5min.
Extracción de ADNEl ADN total se extrajo de cada muestra mediante el procedimiento descrito por Martínez et al.15 con las modificaciones publicadas por Tapia-Paniagua et al.16. Se trataron 20μl de ADN con 2μl de acetato de sodio 3M y 46μl de isopropanol. Las muestras se centrifugaron a 12.000g durante 6min. Los precipitados de ADN se lavaron con etanol al 70% y centrifugados a 12.000 g durante 6 minutos, posteriormente se dejaron secar y fueron resuspendidos en agua químicamente pura.
PCR-electroforesis en gel con gradiente desnaturalizantePara comparar los patrones de electroforesis en gel con gradiente gradiente desnaturalizante (DGGE) de la microbiota duodenal se amplificó el ADN mediante los cebadores específicos para el ADNr16S del dominio Bacteria (968-GC-F y 1401-R). Estos cebadores se utilizaron para amplificar las regiones V6-V8 del ADNr16S. Las mezclas y las condiciones utilizadas para llevar a cabo la PCR se han descrito previamente12. También se emplearon amplicones específicos para el género Lactobacillus (tabla 1). Las mezclas (50μl) contenían 1,25U de polimerasa Taq (Life Technologies Gaithersburg, MD, EE.UU.), 20mM de Tris-HCl (pH8,5), 50mM de KCl, 3mM de MgCl2, 200μM de cada deoxinucleósido trifosfato, 5pmol de los cebadores, 1μl de ADN molde y agua esterilizada mediante radiación ultravioleta. La PCR se llevó a cabo en un termociclador T1 (Whatman Biometra, Göttingen, Alemania) aplicando un ciclo de 2min a 94°C, 35 ciclos de 30s a 95°C, 40s a 56°C, y 1min a 72°C, y finalmente un ciclo de 5min a 72°C. Las alícuotas (5μl) se analizaron mediante electroforesis en gel de agarosa al 1,5% (p/v) que contenía bromuro de etidio.
Los amplicones obtenidos se separaron por DGGE siguiendo las instrucciones de Muyzer et al.17 mediante un sistema Dcode TM (Bio-Rad Laboratories, Hercules, California). La electroforesis se llevó a cabo en gel de poliacrilamida al 8% (37,5:1 acrilamida-bisacrilamida; dimensiones, 200×200×1mm) empleando un gradiente desnaturalizante del 30 al 55% para separar los productos de la PCR. La solución desnaturalizante al 100% contenía 7M de urea y un 40% (v/v) de formamida desionizada. Se añadieron a los geles las muestras de la PCR en alícuotas de 13μl por carril. Se realizó la electroforesis de los geles durante 16h a 85V en tampón TAE 0,5X (20mM tris-acetato [pH7,4], 10mM acetato de sodio, 0,5mM Na2EDTA) a una temperatura constante de 60°C, y a continuación se tiñó con nitrato de plata18.
Análisis de patrones de DGGELos patrones de bandas obtenidos se analizaron mediante el programa FPQuest versión 4.0 (Applied Maths BVBA, Sint-Martens-Latem, Bélgica). Se generó una matriz de similitud para las curvas densitométricas de los patrones de bandas utilizando el coeficiente de Pearson. Posteriormente se agruparon los patrones por similitud mediante la construcción de dendrogramas aplicando el método UPGMA (del inglés, Unweighted Pair Group Method with Arithmetic Mean). Para determinar la diversidad estructural de la comunidad microbiana correspondiente a los patrones de bandas de la DGGE se calcularon varios parámetros: a)la riqueza específica (R) se estimó en base al número total de bandas; b)el índice de Shannon (H′) se calculó mediante la función H′=–ΣPi logPi, en la que Pi se define como (ni/N), ni es el área del pico de cada banda y N es la suma de las áreas de intensidad de todas las bandas; c)la riqueza ponderada por el rango (Rr) se calculó como el número total de bandas multiplicado por el porcentaje de gradiente desnaturalizante requerido para describir la diversidad total de la muestra analizada, de acuerdo con la siguiente fórmula: Rr=(N2×Dg), donde N representa el número total de bandas en el patrón, y Dg el gradiente de desnaturalización comprendido entre las bandas primera y última del patrón19.
Secuenciación de las bandas de DGGEPara determinar cuáles eran las bacterias principales, se recuperaron para su secuenciación las bandas predominantes de los geles de DGGE mediante puntas de pipeta estériles, introduciéndose en 100μl de agua bidestilada (ddH2O) e incubándose a 4°C hasta el día siguiente. Se emplearon alícuotas de 5μl como molde para amplificar el ADN mediante PCR, siguiendo el procedimiento explicado anteriormente. Los productos se volvieron a someter a DGGE para confirmar sus posiciones y también fueron secuenciados por ciclos con los cebadores 968 sin la pinza GC (5′-AACGCGAAGAACCTTAC-3′), 1401-R y (5′ACG’GCTACCTTGTTACGACTT-3′). La PCR se llevó a cabo en un termociclador T1 (Whatman Biometra, Göttingen, Alemania) aplicando un ciclo a 94°C durante 2min, 28 ciclos a 95°C durante 30s, 56°C durante 40s y 72°C durante 1min, y por último un ciclo a 72°C de 5min. Los productos se purificaron con el kit de purificación Spin Kit PCR (Roche). Los amplicones se secuenciaron en un secuenciador ABI PRISM 377 (Perkin-Elmer). La secuenciación se hizo en ambas direcciones con los cebadores RV-M y M13-47, respectivamente. Las secuencias resultantes (∼500pb) se compararon con las secuencias de las bases de datos de la National Center for Biotechnology Information (NCBI) o la Greengenes DNA sequence database empleando el algoritmo BLAST21. Las secuencias mostraron una similitud con las presentes en la base de datos superior al 97%22.
Análisis de componentes principalesEl análisis de componentes principales (ACP) es un método matemático que emplea una transformación ortogonal para convertir un conjunto de observaciones de variables posiblemente correlacionadas en un conjunto de valores de variables linealmente incorrelacionadas entre sí. Este método ha sido empleado para simplificar patrones de RFLP23. En nuestro estudio se aplicó a la correlación de diferentes parámetros ecológicos (H’, R y Rr) e histológicos y la proporción de ADN de cada especie bacteriana detectada en las muestras de la microbiota intestinal de los pacientes. El ACP se realizó con la ayuda del software XLSTAT versión 4.5 para MS Excel (Addinsoft, España). La influencia de las variables en el ACP se determinó mediante la distancia de cada variable al centro del eje d≥√2/n, donde n es el número de variables. El ADN se cuantificó por medio de la intensidad de las bandas de DGGE.
Análisis estadísticoLas variables se expresaron como media y desviación estándar y el análisis estadístico se llevó a cabo con el software Statistical Package for Social Science for Windows (SPSS) versión 15.0, SPSS Inc., Chicago, IL, EE.UU.). Los índices ecológicos se analizaron mediante la prueba de Kruskal-Wallis con el software Statgraphics Plus 5.0 (Statgraphics Corporation, Rockville, MD, EE.UU.). La significación estadística se estableció en p≤0,05.
ResultadosSe recogieron especímenes mediante biopsia duodenal de 11 niños que seguían una dieta con gluten al diagnóstico de EC y de 6 niños sanos (tabla 2)
Características clínicas de los sujetos del estudio: pacientes celíacos sin tratar y controles sanos
| Característica | EC | CS | p |
|---|---|---|---|
| Número | 11 | 6 | |
| Media (DE) edad (años) | 5,0 (3,4) | 8,8 (3,1) | |
| Sexo (masculino/femenino) | 4/7 | 3/3 | |
| Anticuerpos antitransglutaminasa tisular IgA positivos | 11 | 0 | |
| Anticuerpos antigliadina deaminada IgG positivos | 11 | 0 | |
| Anticuerpos antiendomisio IgA positivos | 11 | 0 | |
| Biopsia duodenal: clasificación de Marsh (número de casos) | Marsh 2 (1) Marsh 3a (2) Marsh 3b (2) Marsh 3c (6) | Marsh 0 (6) |
Clasificación de Marsh modificada: Marsh0, mucosa normal; Marsh1, lesión infiltrativa: linfocitosis intraepitelial aumentada en el epitelio de las vellosidades en una mucosa por lo demás normal con una relación vellosidad-cripta normal; Marsh2, tipo hiperplásico: hiperplasia de las criptas con vellosidades normales y linfocitosis intraepitelial aumentada; Marsh3, lesión atrófica con hiperplasia de las criptas y elevación de linfocitos intraepiteliales (Marsh3a: atrofia vellositaria parcial; Marsh3b: atrofia vellositaria subtotal; Marsh3c: atrofia vellositaria total) 14.
No se encontraron diferencias significativas entre sujetos sanos y pacientes celíacos en la riqueza, la diversidad y en la riqueza ponderada por el rango (tabla 3), aunque estos 3 valores fueron significativamente menores en niños celíacos en comparación con CS cuando se analizó el género Lactobacillus (tabla 3).
Riqueza, diversidad y riqueza ponderada por el rango de la microbiota en especímenes obtenidos por biopsia de pacientes celíacos (EC) y controles sanos (CS)
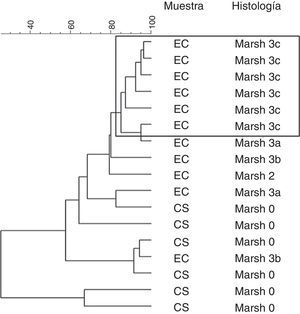
También se aplicó un algoritmo de agrupamiento a los patrones de DGGE de la microbiota duodenal. Esto reveló altos porcentajes de similitud intragrupo (98%) en pacientes con histología Marsh3c. El resto de los pacientes mostró una similitud del 80 al 30%, y el grupo no se formó en base a otras características (fig. 1).
Se hizo un segundo dendrograma empleando exclusivamente los amplicones del género Lactobacillus, que mostró 2 grupos distintos con una similitud del 40% aproximadamente, pero los grupos no se formaron en base a ninguna de las características analizadas (datos no mostrados).
Las bandas de mayor intensidad en los geles de DGGE de las biopsias se secuenciaron y compararon con las referencias de BLAST en base a la relación filogenética con la secuencia parcial de ADNr16S de ∼500pb (tabla 4). En pacientes celíacos, las bandas se asociaban mayoritariamente a géneros como Streptococcus, Bacterioides y la especie E. coli, mientras que en sujetos sanos las bandas predominantes correspondían a Bifidobacterium, Acinetobacter y Lactobacillus. Otros grupos, como Streptococcus (17 al 13%) o Bacteroides (16 al 11%) fueron menos frecuentes.
Abundancia relativa de distintos grupos microbianos secuenciados de la microbiota duodenal de niños con enfermedad celíaca (EC) y controles sanos (CS)
| CS (%) | EC (%) | |
|---|---|---|
| Lactobacillus | 13 | 7 |
| Streptococcus | 13 | 17 |
| Bacteroides | 11 | 16 |
| Acinetobacter | 10 | 0 |
| E. coli | 10 | 16 |
| Bifidobacterium (B. brevis, B. bifidum, B. lactis) | 10 | 5 |
| Clostridium (C. coccoides) | 9 | 8 |
| Eubacterium | 9 | 7 |
| No identificados | 9 | 16 |
| No cultivados | 6 | 8 |
El ACP mostró que 2 factores (F1 y F2) podían explicar el 83,44% de la varianza encontrada en la microbiota y las demás variables analizadas en pacientes y controles. Cabe destacar que las variables relacionadas con la riqueza, la diversidad y la habitabilidad en el género Lactobacillus cambian de manera inversa a las demás variables empleadas en el F1. Además, el F1 separaba al grupo de CS de los pacientes celíacos, de manera que el F1 puede ser un factor relacionado con la presencia o ausencia de enfermedad (fig. 2).
ACP aplicado a la composición de la microbiota de las biopsias y los parámetros analizados en pacientes celíacos y controles sanos (CS). Correlación de las variables analizadas y los componentes principales que explican el 83,44% de la varianza. Distribución de sujetos celíacos y controles sanos a lo largo de los ejes F1 y F2.
No se encontraron diferencias significativas en la diversidad estructural de la comunidad microbiana correspondiente a los patrones de bandas de DGGE entre pacientes celíacos y controles sanos. El estudio de DGGE con cebadores universales no mostró una mayor diversidad bacteriana en la microbiota del intestino delgado de pacientes con EC, pero los índices ecológicos (H’, R y Rr) fueron significativamente más bajos en celíacos que en CS cuando se analizó el género Lactobacillus por separado.
Nadal et al.5 llevaron a cabo análisis bacteriológicos de especímenes de biopsias duodenales realizadas a pacientes pediátricos con EC. Sus resultados mostraron que los pacientes con enfermedad activa tenían un número total de bacterias significativamente mayor, sobre todo de bacterias gramnegativas, en comparación con pacientes asintomáticos y sujetos sanos; y que la razón de Lactobacillus-Bifidobacterium entre Bacteroides-Escherichia coli era más baja en pacientes celíacos. Nistal et al.9 analizaron la secuenciación del gen ARNr16S bacteriano del ADN extraído de biopsias duodenales y mostraron que la diversidad de la microbiota duodenal difería de manera significativa entre adultos celíacos tratados y no tratados.
En nuestro estudio, los pacientes con lesiones Marsh3c se agrupaban por separado en el ACP de los patrones de DGGE. El resto de los pacientes que tenían histologías Marsh3a y Marsh3b y un caso con Marsh2 compartían una microbiota diferente en comparación con los pacientes con Marsh3c, que estaban agrupados más estrechamente. Esto sugiere que la composición de la microbiota duodenal variaba en función del grado de daño intestinal.
Es probable que las condiciones del medio ambiente intestinal en casos de atrofia vellositaria grave causen cambios profundos en la comunidad microbiana. Puede que algunos de los cambios en la composición de la microbiota duodenal se deban a las consecuencias del patrón inflamatorio destructivo, lo que es más evidente en la atrofia vellositaria duodenal más avanzada. En estos casos el efecto inflamatorio del gluten puede incrementar aún más la activación de la inflamación intestinal que resulta del patrón destructivo de lesión atrófica asociado al tipo Marsh3c. Podría pensarse que la atrofia extrema de la mucosa duodenal en la EC no tratada, del tipo Marsh3c, promueve una colonización similar de la mucosa en estos pacientes.
Harán falta más estudios para entender la interacción microbiota-huésped y desentrañar la relevancia de microbiotas específicas para determinar si son únicamente consecuencia de la EC o si están involucradas en la variabilidad y en distintos grados de enteropatía en esta enfermedad. A este respecto, recientemente se han asociado alteraciones en la microbiota, como la reducción de la riqueza microbiana, con la persistencia de síntomas en pacientes celíacos tratados24.
Esperamos que en el futuro la investigación encuentre marcadores bacteriológicos en la microbiota duodenal de pacientes celíacos por razones de diagnóstico y tratamiento.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.