Las causas monogénicas de obesidad constituyen un pequeño porcentaje en el conjunto de la prevalencia de esta enfermedad, de etiología mayoritariamente poligénica1. Sin embargo, es precisamente en la edad pediátrica en la que aquellos casos de obesidades graves de inicio precoz precisan de un mayor esfuerzo diagnóstico, particularmente cuando se asocian a estigmas malformativos1,2. El síndrome de Bardet-Biedl (SBB) (MIM 209900) constituye un ejemplo de una enfermedad clínicamente identificable, pero de difícil diagnóstico molecular. Se conocen hasta 15 subtipos de este síndrome, causados por mutaciones en genes ubicados en otras tantas localizaciones cromosómicas3–5. Además de la presencia de mutaciones en los 2 alelos de uno de esos genes según un patrón autosómico recesivo, existen casos de SBB en quienes se detecta una tercera mutación en otro de los genes candidatos que puede actuar como modificador del fenotipo con un modelo de herencia trialélica5. Dada esta heterogeneidad genética, el estudio mediante secuenciación del exoma (porción codificante del genoma) constituye una buena opción, frente a la secuenciación secuencial de los distintos «genes candidatos», para la optimización de los recursos técnicos y económicos disponibles en el diagnóstico etiológico de muchas enfermedades con patrón de herencia mendeliano en pediatría6,7.
Presentamos el caso de un niño de 2 años y 7 meses de edad, cuyos padres (etnia árabe, no obesos y consanguíneos en segundo grado) solicitaron estudio por obesidad mantenida desde el antecedente de macrosomía neonatal (peso: 4.200 g), pese a una ingesta alimentaria referida como adecuada. Entre los antecedentes personales, destacaba la existencia de un retraso en la adquisición de los hitos del desarrollo psicomotor (deambulación a los 24 meses), más acentuado en el área del lenguaje (mínimos bisílabos referenciales) y una polidactilia (6 dedos) presente en las 4 extremidades y detectada al nacimiento.
En la exploración física: índice de masa corporal de 23,3kg/m2 (+5,63 DE), talla de 95cm (p90); con abundante panículo adiposo de distribución generalizada, pero predominio abdominal. No se objetivaron alteraciones de la pigmentación de la piel ni del cabello, pero se comprobó la existencia de polidactilia postaxial en los 4 miembros (apéndice rudimentario en la mano derecha), con sindactilia parcial entre el 5.° y el 6.° dedos de ambos pies (malformaciones presentes también en un primo materno). Asimismo, destacaba la presencia de nistagmo vertical bilateral, pene de 2×1cm, fino y enterrado en el tejido adiposo y escaso desarrollo de la bolsa escrotal, aunque con testes de 1 cc de volumen y consistencia normal localizados en la misma.
Las exploraciones complementarias practicadas (hemograma, bioquímica sérica general, niveles circulantes de tirotropina, tiroxina libre, insulinemia y perfil lipídico) fueron normales; así como la edad ósea, el cariotipo —46 XY—, el estudio cardiológico y la ecografía renal. El estudio radiológico del esqueleto no mostró alteraciones, mientras que el estudio oftalmológico objetivó distrofia retiniana bilateral (retinitis pigmentaria), miopía magna y catarata polar posterior en ojo derecho.
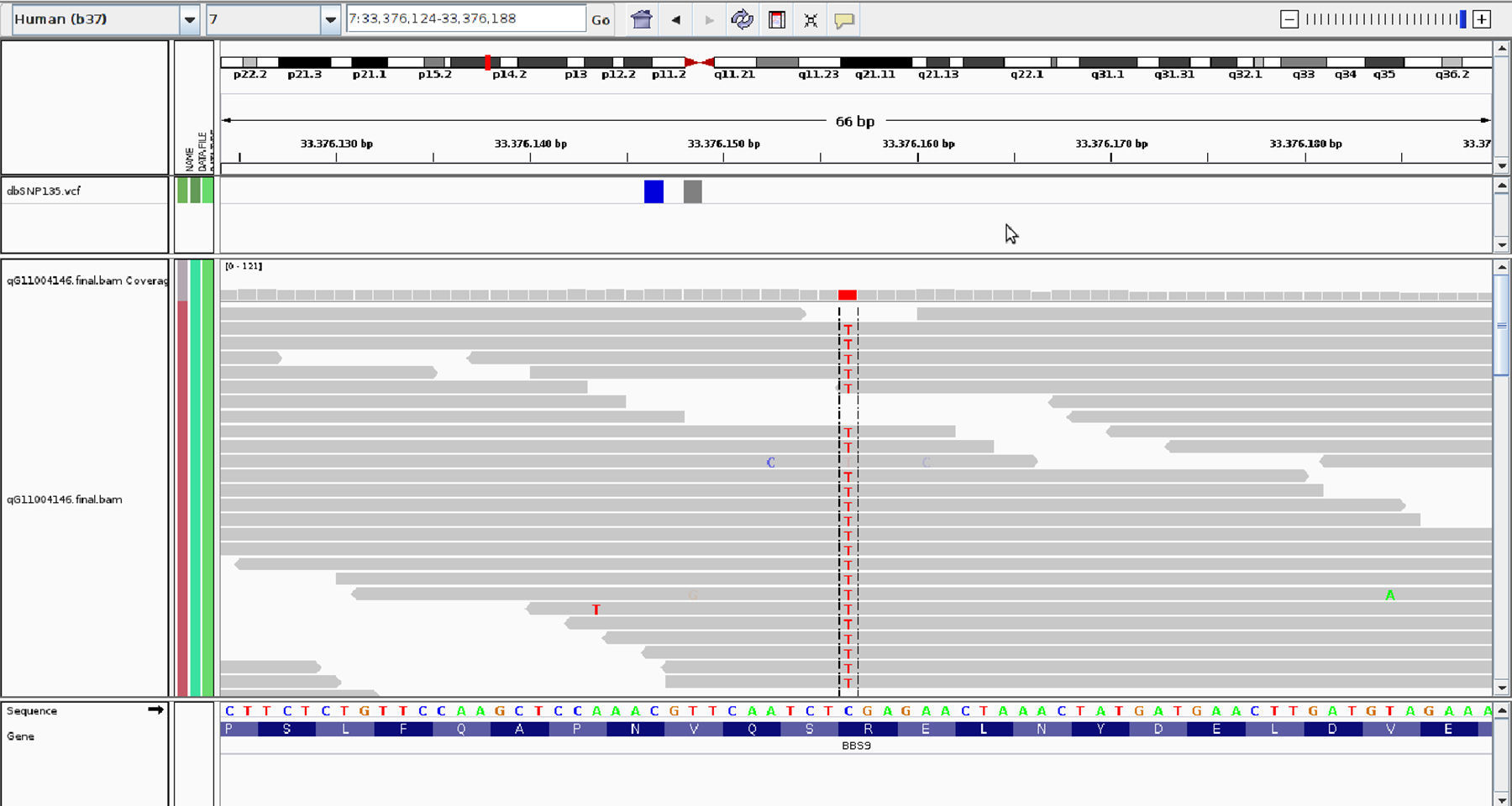
Se estableció la sospecha clínica de SBB. Se optó por solicitar el estudio de una muestra de ADN mediante secuenciación del exoma (qGenomics, Barcelona) (fig. 1). Se identificó una mutación sin sentido (c.C1120T; p.R374X) en homocigosis, en el gen PTHB1/BBS9 (7p14.3), que determina la formación de un codón de parada prematuro, interrumpiendo la traducción de la proteína BBS9, imprescindible en el proceso de la ciliogénesis8. Las mutaciones del gen PTHB1/BBS9 han sido identificadas como las responsables del subtipo número 9 del SBB. Además se detectó una tercera mutación de mal sentido (c.G1799A; p.R600H) en el exón 13 del gen WDPCP/C2orf86/BBS15 que origina un cambio funcional en la proteína WDPCP, implicada en el control de la polaridad celular, que podría constituir el alelo modificador en un modelo trialélico. Por tanto, la secuenciación del exoma consiguió la confirmación de la base molecular de la enfermedad de manera rápida (6 semanas) y con la máxima eficiencia.
Imagen extraída del estudio de exoma, visualizada en el visor integrado del genoma (IGV, http://www.broadinstitute.org/igv/), en la que se muestra la sustitución detectada (citosina por timina) en todas las lecturas de secuencia y la localización cromosómica de la misma (marca vertical de color rojo en el esquema del cromosoma 7 en la parte superior de la figura.
La confirmación molecular del diagnóstico clínico es imprescindible para facilitar el asesoramiento genético con la posibilidad de ofertar diagnóstico prenatal o preimplantatorio para futuras gestaciones de los padres del probando.
Este caso clínico ilustra la necesidad de plantear la eventual etiología monogénica (sindrómica o no) en aquellos casos de obesidad grave de inicio precoz, particularmente en los pacientes que, como este, exhiben estigmas dismórficos asociados. Las herramientas de «secuenciación masiva» como el estudio del exoma pueden suponer, frente a la secuenciación individualizada y sucesiva de diversos genes candidatos, un considerable ahorro en el tiempo y en los recursos invertidos en el estudio, acelerando así el establecimiento del diagnóstico y optimizando el empleo de recursos económicos.