La etiología de la enfermedad de Kawasaki (EK) sigue siendo desconocida. Varios estudios han relacionado el microbioma humano con algunas enfermedades. Sin embargo, los estudios sobre el microbioma respiratorio en EK son limitados. Este estudio intenta profundizar en las causas y procesos que predisponen al desarrollo de la EK.
MétodosEstudio de casos y controles en el que se compara el microbioma respiratorio de pacientes con EK con el de niños sanos. La región V3-V4 del gen bacteriano del ARNr 16S y 16 virus respiratorios se analizaron mediante reacción en cadena de la polimerasa en tiempo real. Se utilizó la base de datos RDP (Ribosomal Database Project) versión 11.5 (asignación taxonómica).
ResultadosSe incluyeron 11casos y 11 controles emparejados por edad, sexo y estacionalidad. Uno de los casos fue descartado por mala calidad de la muestra. El estudio final se realizó a 10 casos y 10 controles. En el grupo de casos se encontraron Haemophilus, Moraxella, Streptococcus y Corynebacterium (27,62%, 19,71%, 25,28% y 11,86%, respectivamente). En el grupo control, Haemophilus, Streptococcus, Moraxella y Dolosigranulum (38,59%, 23,71%, 16,08 y 8,93%, respectivamente). Corynebacterium mostró una mayor abundancia en pacientes con EK (11,86% vs. 1,55%; p = 0,004).
ConclusionesHasta donde sabemos, este es el primer estudio que ha encontrado diferencias en la composición del microbioma respiratorio entre pacientes con EK y controles sanos. Corynebacterium spp. presentó una mayor abundancia en el grupo de EK. Este estudio muestra diferencias en el microbioma entre pacientes y controles, lo que sugiere un papel facilitador del microbioma en el desarrollo de la EK.
The aetiology of Kawasaki disease (KD) remains unknown. Several studies have linked the human microbiome with some diseases. However, there are limited studies on the role of the respiratory microbiome in KD. The aim of our study was to make a more thorough analysis of the causes and processes that increase the susceptibility to KD.
MethodsCase-control study comparing the respiratory microbiome of KD patients with that of healthy children. The V3–V4 region of the 16S rRNA bacterial gene and 16 respiratory viruses were analysed by real-time polimerase-chain reaction. We used the Ribosomal Database Project (RDP) version 11.5 (taxonomic assignment).
ResultsThe initial sample included 11 cases and 11 controls matched for age, sex and seasonality. One of the cases was excluded to poor sample quality. The final analysis included 10 cases and 10 controls. In the case group, the analysis detected Haemophilus, Moraxella, Streptococcus and Corynebacterium species (27.62%, 19.71%, 25.28%, 11.86%, respectively). In the control group, it found Haemophilus, Streptococcus, Moraxella, and Dolosigranulum species (38.59%, 23.71%, 16.08, 8.93%, respectively). We found a higher relative abundance of Corynebacterium in patients with KD (11.86% vs. 1.55%; P=.004).
ConclusionsTo our knowledge, this is the first study that has found differences in the composition of the respiratory microbiome between patients with KD and healthy controls. The relative abundance of Corynebacterium spp. was greater in the KD group. This study shows differences in the microbiome between cases and controls, which suggests that the microbiome may play a role in facilitating the development of KD.
La enfermedad de Kawasaki (EK) es una vasculitis sistémica, aguda y autolimitada de la infancia que afecta principalmente a lactantes y niños menores de 5 años. Su diagnóstico es clínico. Su pronóstico depende de la afectación cardíaca. Sin tratamiento, del 20 al 25% de los pacientes desarrollan aneurismas coronarios1.
La etiología de la EK se desconoce a pesar de años de investigación. Los hallazgos clínicos, de laboratorio y las características epidemiológicas de la enfermedad señalan un origen o desencadenante infeccioso. Hasta la fecha, numerosos estudios han fracasado en el intento de identificar un agente etiológico infeccioso o evidenciar una asociación con la exposición a medicamentos o la respuesta a un superantígeno. También se ha demostrado una importante predisposición genética a la EK. Algunos estudios de asociación de genoma completo (GWAS) han identificado varios loci biológicamente plausibles, implicados en la inflamación, la respuesta inmunitaria y el estado cardiovascular2-4. En función del agrupamiento espacial o temporal de casos, entre otros patrones, una hipótesis razonable es que la EK podría ser causada por un desencadenante (debatiéndose aún si sería un agente infeccioso) que solo produciría enfermedad o generaría una respuesta inmunológica en individuos con predisposición genética, especialmente de origen asiático1. En cambio, se sabe poco del impacto del microbioma humano en el desarrollo de la EK.
El término microbioma hace referencia a la comunidad de microorganismos comensales, simbióticos y patogénicos que comparten el espacio corporal humano5. Varios estudios han relacionado el microbioma humano con algunas enfermedades o la predisposición a desarrollarlas. Algunos de ellos han investigado la asociación entre la desregulación del microbioma intestinal y múltiples enfermedades autoinmunes. Los modelos animales sugieren que el microbioma intestinal contiene organismos que dirigen respuestas inmunitarias proinflamatorias y antiinflamatorias según el contexto inmunológico, el genotipo o el sexo de huésped y la estructura general de la comunidad microbiana6.
En el campo de la Reumatología, el papel del microbioma humano se ha estudiado en enfermedades como la artritis reumatoide (AR) o el lupus eritematoso sistémico (LES). En el caso de la AR, se ha encontrado una asociación entre un aumento en la presencia de Porphyromonas encialis, un anaerobio oral involucrado en el desarrollo de la periodontitis y resultados positivos de anticuerpos antipéptido citrulinado cíclico7. También se han detectado alteraciones de la microbiota intestinal en la AR, con una expansión considerable de Prevotella copri en pacientes sin tratar con AR de inicio reciente8. El LES también se caracteriza por alteraciones microbianas. Uno de los primeros estudios encontró una diversidad bacteriana reducida y una ratio de Firmicutes y Bacteroidetes inferior en pacientes con LES9. Al igual que en la AR, hay evidencia de una translocación de bacterias intestinales en el LES, con detección de Enterococcus gallinarum en biopsias hepáticas de pacientes con LES activo10.
Aunque el papel de la desregulación del microbioma intestinal en la patogénesis de las enfermedades autoinmunes se ha estudiado extensamente, el papel del microbioma respiratorio en la autoinmunidad no está tan claramente establecido. Aunque no se ha identificado ningún agente causal infeccioso, algunos estudios epidemiológicos han descrito infecciones por diversos microorganismos en muchos pacientes con EK. Recientemente se han descritos casos con una presentación similar a la de la EK en asociación a la infección por SARS-CoV-211,12. Por añadidura, el hecho de que ha habido epidemias de EK en Japón, su mayor incidencia en primavera e invierno y su ocurrencia predominantemente en niños de 6 meses a 5 años de edad apoyan la hipótesis de una etiología o desencadenante infeccioso1.
Hasta donde conocemos, los estudios sobre el microbioma respiratorio en la EK son limitados. Algunos estudios han buscado una relación entre la flora intestinal y faríngea con la EK y no encontraron diferencias significativas entre casos y controles13,14. En un estudio del microbioma intestinal en EK publicado en 2005 se analizó el microbioma fecal de 28 pacientes con EK, objetivándose un aumento en las especies de Streptococcus en la fase aguda de la enfermedad y en las bacterias del género Ruminococcus en la fase subaguda15. Avances recientes permitieron descubrir que la diferenciación de las células Treg y Th17 está regulada por ácidos grasos de cadena corta (AGCC), en particular el butirato, producidos por la microbiota intestinal. Este hallazgo apuntó a una relación mecanística entre la disbiosis, definida como cambios en la composición de la microbiota intestinal, y varias enfermedades inflamatorias. Estudios recientes han intentado corroborar esta hipótesis, objetivándose concentraciones fecales de butirato significativamente disminuidas en pacientes con EK, lo que supone una información importante en cuanto a la asociación entre la disbiosis y la respuesta inmune desregulada en la EK15-17.
Hallazgos ultraestructurales han sugerido la contribución de los virus a la patogénesis de la EK, habiéndose encontrado cuerpos de inclusión citoplasmáticos que contenían ARN de origen viral en el epitelio bronquial de pacientes con EK fallecidos18-20. Otros estudios han encontrado virus respiratorios en muestras de aspirado nasofaríngeo en aproximadamente la mitad de los pacientes con EK21.
Algunos trabajos han apuntado a una posible contribución de infecciones fúngicas en el desarrollo de la EK. Estudios recientes22,23 indican que el agente causal podría ser un agente ambiental transportado en vientos troposféricos. Sus autores encontraron toxinas aerosolizadas de varias especies de Candida en muestras recogidas de la troposfera en periodos de alta incidencia de EK. En Japón, el número de casos frecuentemente parece estar asociado con corrientes de aire provenientes de terrenos de cultivo intensivo de cereales en el noreste de China24,25. Por otro lado, Sato et al. observaron que sustancias derivadas de Candida albicans, como su fracción soluble en agua, inducían arteritis coronaria en ratones26.
La mayoría de los hallazgos publicados muestran que la composición del microbioma intestinal en pacientes con EK difiere de la de sujetos sanos16. Una hipótesis razonable para la patogénesis de la EK sería que, en individuos con predisposición genética y un microbioma favorable, un desencadenante externo podría facilitar el desarrollo de la enfermedad. Que conozcamos, ningún estudio ha caracterizado el microbioma encontrado en secreciones respiratorias en la EK.
El objetivo principal del estudio fue comparar las diferencias en la microbiota bacteriana nasofaríngea y en los virus respiratorios en pacientes con EK y controles sanos como primer paso para profundizar en el papel del microbioma y los antecedentes infecciosos en la patogénesis de EK.
Materiales y métodosSe realizó un estudio de casos y controles incluyendo a niños diagnosticados de EK (casos) tratados en un hospital terciario (n = 11) emparejados con niños sanos (controles) atendidos en el mismo hospital que fueron tratados mediante pruebas de laboratorio antes de una cirugía menor o que acudieron a visitas de seguimiento en pediatría general (n = 11). Uno de los casos acabó excluyéndose debido a la baja calidad de la muestra. La muestra final incluyó 10 casos y 10 controles. Las muestras se recogieron entre febrero del 2016 y mayo del 2018. Los casos y controles se emparejaron por edad, sexo y estacionalidad. Para más información sobre la metodología, consultar el material suplementario.
Todos los pacientes se reclutaron en los 5 días que siguieron al diagnóstico y las muestras respiratorias se tomaron antes de iniciarse el tratamiento. Todos cumplieron los criterios diagnósticos de EK establecidos por la American Heart Association1. Se excluyó a pacientes que habían recibido antibioterapia antes de la toma de la muestra o para los que no se obtuvo el consentimiento informado por escrito. Los controles eran niños sanos sin comorbilidades importantes y que no habían recibido tratamiento antibiótico en las 4 semanas que precedieron a la toma de la muestra. Los conjuntos de datos presentados en este artículo se pueden consultar en repositorios en línea. El nombre del o los repositorios y el número o los números de acceso se detallan en el repositorio de Figshare dentro del proyecto Kawasaki: https://figshare.com/s/181b728556419d0de4c5.
Obtención de muestras de aspirado nasofaríngeoSe obtuvo una muestra de aspirado nasofaríngeo de cada paciente para su análisis. Para ello, se inyectaron de 2 a 3ml de cloruro de sodio Meinsol® 9mg/ml (Fresenius Kabi; Alemania) en la nasofaringe del paciente, para luego aspirar la solución y almacenarla en tubos estériles de 25ml. Se almacenaron alícuotas en tubos Eppendorf de 1,5lL (STARLAB International GmbH, Hamburgo, Alemania), que se congelaron de inmediato a –80°C. También se procesaron 6 muestras de control (2-3ml de cloruro de sodio Meinsol 9 mg/ml disolvente para uso parenteral [Fresenius Kabi España, S.A.U.]). Tanto las muestras clínicas como las de control fueron tratadas mediante el mismo protocolo de recogida, extracción, amplificación y secuenciación de ADN. Las muestras se procesaron en un laboratorio de diagnóstico clínico en áreas libres de ácidos nucleicos y en cabinas con flujo laminar tratadas con rayos UV, con reactivos y pipetas dedicados exclusivamente a este proceso.
Extracción de ADN/ARNLa extracción de ADN de muestras de aspirado y controles negativos se realizó con el sistema Nuclisens® EasyMag® de acuerdo con las instrucciones del fabricante (bioMérieux S.A. 69280 Marcy l’Etoile/France). Es un sistema automatizado de extracción de ácidos nucleicos basado en columnas de sílice con partículas magnéticas. Partiendo de una muestra con un volumen de 400μl, se obtenía una elución con un volumen de 25μl. A continuación, el ADN extraído se cuantificaba por medio del Qubit dsDNA HS Assay Kit (Thermo Fisher Scientific, Massachusetts, EE. UU.), un método fluorométrico basado en la unión específica de colorantes fluorescentes al ADN bicatenario. La extracción de ADN/ARN para la detección de virus respiratorios se realizó con el sistema Magna Pure (Roche Diagnostics, Indianápolis, IN, EE. UU.), otro sistema de extracción automatizado basado en partículas magnéticas de sílice, siguiendo las instrucciones del fabricante.
Estudio del microbioma mediante secuenciación de nueva generación (NGS)La región V3-V4 del gen de la subunidad 16S del ARN ribosomal (ARNr 16S), de unos 465 pares de bases (pb), se amplificó mediante reacción en cadena de la polimerasa (PCR) utilizando cebadores específicos y con las siguientes condiciones de amplificación: 95°C (3min), seguido de 25 ciclos a 95°C (30 s), 55°C (30 s), 72°C (30 s) y 72°C (5min). A continuación, se procedió a fragmentar el ADN total de cada muestra, añadiéndose 2índices y adaptadores de secuenciación con el Nextera XT Illumina Index Kit (Illumina, San Diego, California, EE. UU.) mediante PCR. Se realizó una segunda purificación con esferas magnéticas (Agencourt AMPure XP 60ml kit, Beckman Coulter; Múnich, Alemania) antes de pasar a la cuantificación de la genoteca con el Qubit dsDNA HS Assay Kit (Thermo Fisher Scientific, Massachusetts, EE.UU.). Se construyó una genoteca de clones de 300 pb por muestra. Se mezclaron cantidades equimolares de ADN de cada muestra y el conjunto resultante se secuenció con el sistema Illumina MiSeq (Illumina; San Diego, California, EE. UU.).
Detección de virus respiratoriosSe utilizó una prueba de reacción en cadena de la polimerasa en tiempo real (RT-PCR) multiplex (Allplex Respiratory Panel Assays) para la detección de 16 virus respiratorios y 3 subtipos de influenza A que causan infecciones de las vías aéreas: influenza A (subtipos H1, H1pdm09 y H3), influenza B, virus respiratorio sincitial A y B, adenovirus, enterovirus, parainfluenzavirus 1, 2, 3 y 4, metapneumovirus, bocavirus, rinovirus, coronavirus NL63, coronavirus 229E y coronavirus OC43.
Análisis estadísticoSe evaluó la normalidad de los datos mediante la prueba de Shapiro-Wilk test. Los datos cuantitativos con distribución normal se describieron mediante la media y la desviación estándar (DE). Los grupos se compararon mediante la prueba de la t de Student en el caso de seguir una distribución normal y de lo contrario mediante la U de Mann-Whitney. Para la comparación de más de 2grupos, se utilizó la prueba de Kruskal-Wallis. El contraste de hipótesis en variables cualitativas se realizó con el test exacto de Fisher. Los valores de p inferiores a 0,05 se consideraron estadísticamente significativos.
Consideraciones éticasEl estudio fue aprobado por el Comité Ético del hospital. Todos los procedimientos se llevaron a cabo de acuerdo con las guías y legislación pertinentes, y se obtuvo el consentimiento informado de todos los participantes o sus tutores legales. El estudio se realizó en conformidad con los principios de la Declaración de Helsinki.
ResultadosCaracterísticas de los pacientesLa muestra inicial incluyó a 11 pacientes con EK (6 varones y 5 mujeres), de los que uno fue excluido debido a la baja calidad de la muestra; los pacientes tenían de 6 a 81 meses de edad (mediana, 30 meses). Todos los pacientes cumplían los criterios diagnósticos de EK establecidos por la American Heart Association1. Todos los pacientes con EK recibieron inmunoglobulina por vía intravenosa (IGIV) en dosis de 2g/kg como tratamiento inicial y solo 2 requirieron tratamiento adicional, como una segunda dosis de IGIV o corticoides. Las características clínicas y el tratamiento de los pacientes se presentan en la tabla 1. Los casos y los controles se emparejaron por edad (año de nacimiento), sexo y estacionalidad (misma estación). Los controles eran niños sanos sin comorbilidades que acudieron al mismo hospital por seguimiento en pediatría general o previamente a una cirugía menor.
Características de los pacientes con enfermedad de Kawasaki al ingreso
| Paciente | Sexo | Edad | Fecha de inicio | Fecha de obtención de muestra | Subtipo de EK | Síntomas concomitantes | Afección coronaria al ingreso | Leucocitos totales (/mm3) | Plaquetas totales (/mm3) | Sodio (mmol/l) | VSG (mm/h) | PCR (mg/l) | Respuesta a la IGIV |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| P1 | V | 1 a 2 m | 26 enero, 2016 | 2 febrero, 2016 | I | Diarrea, vómitos | No | 15.100 | 321.000 | 138 | 33 | 47,7 | Sí |
| P2 | V | 1 a 10 m | 1 febrero, 2016 | 8 febrero, 2016 | C | – | No | 23.300 | 849.000 | 133 | 44 | 24,8 | Sí |
| P3 | V | 6 a 6 m | 12 abril, 2016 | 22 abril, 2016 | C | No | 13.100 | 507.000 | 137 | ND | 17,8 | Sí | |
| P4 | M | 7 m | 14 abril, 2017 | 19 abril, 2017 | C | Piuria estéril | No | 20.500 | 586.000 | 136 | ND | 110,3 | Sí |
| P5 | M | 3 a 1 m | 24 abril, 2017 | 29 abril, 2017 | C | Piuria estéril | No | 17.300 | 343.000 | 137 | 23 | 79,3 | No |
| P6 | V | 6 m | 3 julio, 2017 | 7 julio, 2017 | C | – | No | 19.800 | 449.000 | 136 | 54 | 301,5 | Sí |
| P7 | V | 3 a 8 m | 5 julio, 2017 | 9 julio, 2017 | C | Vómitos | No | 7.100 | ND | 130 | ND | 161,0 | Sí |
| P8 | M | 9 m | 4 julio, 2017 | 11 julio, 2017 | I | Diarrea | Sí | 30.900 | 636.000 | 134 | 32 | 204,0 | Sí |
| P9 | M | 2 a 3 m | 30 septiembre, 2017 | 9 octubre, 2017 | I | Hepatomegalia | No | 36.200 | 440.000 | 137 | 42 | 146,1 | Sí |
| P10 | M | 3 a 8 m | 18 mayo, 2018 | 24 mayo, 2018 | C | SAM | No | 3.000 | 154.000 | 138 | ND | 279,3 | No |
a: años; C: completa; I: incompleta; IGIV: inmunoglobulina por vía intravenosa; m: meses; M: mujer; ND: no documentado; PCR: proteína C reactiva; SAM: síndrome de activación macrofágica; V: varón; VSG: velocidad de sedimentación globular.
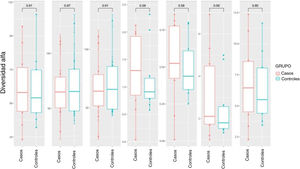
La diversidad alfa se centra en el estudio de la diversidad dentro de una única comunidad (intramuestra). Para su medida se utilizan distintos parámetros, cada uno de los cuales contribuye a explicar uno o más se sus componentes. Dentro del análisis de la diversidad alfa, se puede diferenciar entre la riqueza y la diversidad. La riqueza solo tiene en cuenta el número de géneros distintos contenidos en cada una de las muestras, mientras que la diversidad también tiene en cuenta la abundancia de estos géneros en las muestras. Se analizaron distintos índices de diversidad alfa tanto en casos como en controles, representándose por medio de diagramas de cajas (fig. 1). No se encontraron diferencias significativas ni en la riqueza ni en la diversidad.
Diversidad alfa: diagrama de caja de los distintos índices de diversidad y riqueza en los grupos de casos y de control. La línea horizontal en cada caja representa la mediana del conjunto de datos, los bordes de la caja los percentiles 25 y 75. El valor de p obtenido mediante la prueba de Wilcoxon se muestra encima de la caja.
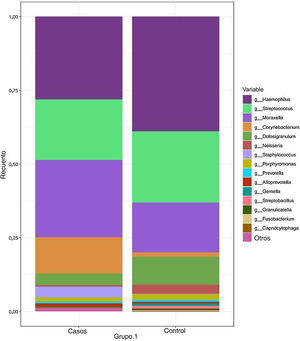
Los géneros más frecuentes en el grupo de casos fueron Haemophilus, Streptococcus, Moraxella y Corynebacterium, con abundancias relativas del 27,55, el 20,62, el 26,20 y el 12,25%, respectivamente. En comparación, el género dominante en el grupo control fue Haemophilus, seguido de Streptococcus, Moraxella y Dolosigranulum (el 38,83, el 24,21, el 16,79 y el 9,40%, respectivamente). La abundancia relativa del resto de los géneros identificados fue inferior al 4% (fig. 2) (tabla 2).
Composición taxonómica bacteriana de las muestras nasofaríngeas en los grupos de casos y de control: diagrama de barras apiladas que muestra la abundancia relativa (promedio) de los géneros bacterianos detectados con mayor frecuencia en cada grupo. Los géneros con un promedio de abundancia relativa inferior al 0,5% se incluyeron en la categoría «otros géneros».
Abundancias relativas de los géneros detectados con mayor frecuencia en casos y controles
| Género | Casos | Controles | P (Bonferroni-Holm) |
|---|---|---|---|
| Haemophilus | 27,98 ± 34,28 | 38,83 ± 36,60 | 0,696 |
| Streptococcus | 20,62 ± 21,83 | 24,21 ± 27,79 | 1,223 |
| Moraxella | 26,20 ± 33,99 | 16,79 ± 23,49 | 0,607 |
| Corynebacterium | 12,25 ± 22,11 | 1,59 ± 4,63 | 0,045 |
| Dolosigranulum | 3,91 ± 6,46 | 9,40 ± 26,27 | 0,606 |
| Neisseria | 0,604 ± 1,44 | 3,22 ± 8,77 | 0,621 |
| Staphylococcus | 3,59 ± 7,48 | 0,08 ± 29,05 | 0,727 |
| Gemella | 0,17 ± 0,18 | 0,88 ± 0,12 | – |
| Streptobacillus | 0,006 ± 0,012 | 0,95 ± 2,75 | – |
| Granulicatella | 0,12 ± 0,28 | 0,56 ± 1,32 | – |
| Otros | 1,34 ± 2,48 | 0,17 ± 0,33 | – |
Datos expresados como media ± desviación estándar.
Corynebacterium spp. estaba sobrerrepresentado en el grupo con EK (12,25% vs. 1,59%; p = .045). También se observó una proporción mayor de otros géneros con abundancias relativas inferiores a 0,5% en el grupo de casos en comparación con el grupo de control (16,07% vs. 7,82%; p = 0,08).
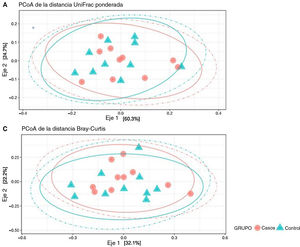
Análisis de diversidad betaLa diversidad beta se define como la diferencia en la diversidad de las especies entre comunidades (intermuestra) y puede revelar aspectos de la ecología microbiana que pueden pasar desapercibidos mediante la mera consideración de la composición individual de las muestras. Para analizar la diversidad beta, se transformaron las unidades taxonómicas operativas (OTU, por sus siglas en inglés) en abundancias relativas, excluyéndose aquellos OTU que correspondían a abundancias relativas inferiores al 0,1%. No se observaron diferencias significativas en las matrices de distancias empleadas (p = 0,61) (fig. 3).
Diversidad beta: las unidades taxonómicas operativas (OTU) son grupos (clústeres) de variantes secuencia del gen ARNr 16S. Cada clúster supuestamente representa una unidad taxonómica de especies, géneros, grupos o taxones. Dependiendo del umbral de identidad entre las secuencias de un clúster, cabe suponer que la OTU corresponde a una especie o género específico. Las OTU con una identidad del 97% corresponden a géneros bacterianos. Las OTU se clasificaron en distintos rangos taxonómicos (dominio, división, orden, familia y género) con un umbral inferior de confianza de 80%. Las OTU se transformaron a abundancias relativas, excluyéndose aquellas correspondientes a una abundancia relativa inferior a 0,1%. No se encontraron diferencias significativas en ninguna de las matrices de distancia empleadas (p = 0,61).
En 80% de los casos de EK (n = 8), la prueba RT-PCR detectó la presencia de ADN/ARN de algunos de los virus respiratorios incluidos en el panel, en comparación con el 60% de los controles sanos (p = 0,659). Los virus detectados con mayor frecuencia en el grupo de EK fueron rinovirus (n = 4; 40%), adenovirus (n = 3; 30%), virus respiratorio sincitial (n = 2; 20%) y parainfluenza 4 (n = 1; 10%), mientras que en el grupo control fueron rinovirus (n = 4; 40%), coronavirus (n = 3; 30%), adenovirus (n = 2; 20%) y bocavirus (n = 1; 10%). No se encontraron diferencias significativas entre los grupos (tabla 3).
Frecuencia de infección viral de vías respiratorias y distribución individual de virus analizada mediante RT-PCR en casos y controles
| Casos (n = 10) | Controles (n = 10) | p (Fisher) | |
|---|---|---|---|
| Infección de vías respiratorias | 8 (80%) | 6 (60%) | 0,659 |
| Virus respiratorio sincitial | 2 (20%) | – | 0,476 |
| Parainfluenza 4 | 1 (10%) | – | 1,000 |
| Adenovirus | 3 (30%) | 2 (20%) | 1,000 |
| Coronavirus | 1 (10%) | 3 (30%) | 0,586 |
| Bocavirus | – | 1 (10%) | 1,000 |
| Rinovirus | 4 (40%) | 4 (40%) | 1,000 |
| Coinfección viral de vías respiratorias (> 1 virus) | 3 (30%) | 3 (30%) | 1,000 |
Hasta donde conocemos, este es el primer estudio que ha encontrado diferencias en la composición del microbioma respiratorio entre pacientes con EK y controles sanos. En 2004, Horita et al. recogieron frotis nasofaríngeos en 21 pacientes con EK y 20 controles con síndrome febril no relacionado con EK. Se aislaron 61 cepas bacterianas de los pacientes con pacientes con EK y 62 cepas de los controles. Los autores no encontraron diferencias entre los pacientes con EK y los controles febriles en las especies bacterianas presentes en la flora nasofaríngea13.
En nuestro estudio, se tomaron muestras de aspirado nasofaríngeo de 10 pacientes con EK y 10 controles sanos y se estudió el microbioma por medio de NGS. Se observó una diferencia en la proporción de Corynebacterium, que estaba sobrerrepresentado en el grupo con EK (12,25% vs. 1,59%; p = 0,045). Además, la proporción de otros géneros con abundancias inferiores al 0,5% también fue mayor en el grupo de casos en comparación con el de control (16,07% vs. 7,82%; p = 0,08). Un estudio previo cuyo objeto era aislar el gen bacteriano del agente causativo de leucocitos periféricos en pacientes con EK identificó una nueva especie del género Corynebacterium27. También se identificaron Corynebacterium spp. en muestras de aerosoles recogidas al nivel del suelo cerca de Tokio en un momento en que la incidencia de EK era alta. Curiosamente, un número importante de taxones adicionales previamente asociados a la EK en distintos estudios también han sido identificados en el análisis metagenómico de muestras de aire. Esto sugiere que la exposición a aire con diferentes cargas bacterianas podría contribuir a la patogénesis de la EK, un aspecto que merece estudiarse con más detalle24,25,28.
El género Corynebacterium es un taxón de bacilos o cocobacilos grampositivos de morfología corineforme29,30. Casi todos son catalasa positivos y expresan un amplio abanico de pigmentos y procesos metabólicos. Las bacterias de este género son ubicuas y se encuentran comúnmente en la piel y mucosas de los seres humanos, y casi todas son saprofitas. Distintas especies de Corynebacterium se han identificado en el oído medio de niños con otitis media, pudiendo causar infecciones oportunistas, y algunas especies, como Corynebacteriumdiphtheriae, son patogénicas para los seres humanos31.
Otros estudios del microbioma intestinal han observado que el género beneficioso Lactobacillus puede estar ausente o desaparecer de la flora enteral durante la fase aguda de la EK14 y que parece haber un aumento relativo del género Streptococcus en la fase aguda de la enfermedad y del Ruminococcus en la fase subaguda15. En nuestro estudio, no encontramos diferencias en ninguno de estos 3 géneros en las muestras, lo que podría deberse al reducido tamaño muestral o al hecho de que se estudiaron muestras respiratorias en lugar de fecales.
La contribución de los virus a la EK viene sugerida por estudios que han encontrado cuerpos de inclusión citoplasmáticos que contenían ARN viral en el epitelio bronquial de pacientes con EK fallecidos19. Aunque varios estudios han intentado identificar el origen de este ARN viral, no se ha identificado ningún virus, y Rowley et al. concluyeron que podría ser un virus ARN ubicuo aún por identificar que podría producir una infección persistente del epitelio bronquial, contribuyendo así a la etiología de la EK18,20. En otro estudio, Turnier et al. encontraron que casi la mitad (n = 93; 41,9%) de los 192 pacientes con EK en estudio tenían una prueba PCR de detección de virus respiratorios positiva, principalmente para rinovirus o enterovirus21. De manera similar, en nuestro estudio no se encontraron diferencias entre casos y controles, aunque rinovirus y adenovirus, entre otros, fueron identificados en casi la mitad de las muestras en ambos grupos.
Aunque nuestro estudio adolece de algunas limitaciones obvias, principalmente el pequeño tamaño muestral y la falta de comparación, no solo con controles sanos, sino también con niños con enfermedades respiratorias, los resultados resultan intrigantes si se contempla la posibilidad de una etiología única para esta enfermedad.
No obstante, es posible que la EK sea una enfermedad multifactorial y que la exposición a una variedad de agentes microbianos o la dominancia de agentes microbianos con similitudes estructurales provoquen la misma respuesta inmunitaria idiosincrática. Hay un nivel de evidencia razonable de que un desequilibrio en el microbioma contribuye a la patogénesis de la EK. Nuestro estudio presenta el género Corynebacterium como un nuevo agente que podría estar en juego y demuestra la necesidad de realizar estudios epidemiológicos a mayor escala para revelar cambios en el microbioma de los pacientes con EK.
El presente artículo no establece una relación causal, pero sienta las bases para realizar estudios en muestras mayores que contribuyan a esclarecer el papel del género Corynebacterium en la patogénesis de EK. Nuestro grupo ya está recogiendo más muestras de pacientes con EK, controles sanos y pacientes con infecciones respiratorias para intentar obtener resultados más concluyentes.
ConclusiónHasta donde conocemos, este es el primer estudio que ha encontrado diferencias en la composición del microbioma respiratorio entre pacientes con EK y controles sanos. El género Corynebacterium presentó una mayor abundancia en el grupo de EK en comparación con el grupo control. Nuestros hallazgos apoyan la hipótesis de que puede haber un sustrato microbiano respiratorio facilitador del desarrollo de enfermedad en pacientes con predisposición genética. Son necesarios estudios en muestras mayores para evaluar su relevancia y posible asociación con la EK.
FinanciaciónEste trabajo ha sido financiado por el Plan Estatal de I+D+i, Instituto de Salud Carlos III (expediente PI20/00517) y cofinanciado por el Fondo Europeo de Desarrollo Regional (FEDER). Parte de la financiación provino de una beca de la Sociedad Española de Reumatología Pediátrica (SERPE) (2018) y una beca de la Fundació Daniel Bravo Andreu (2016) otorgada al proyecto WINDBIOME. Estas instituciones no se involucraron en el diseño del estudio, la recogida de datos, la decisión de publicar o la redacción del manuscrito.
Conflicto de interesesLos autores declaran que la investigación se llevó a cabo en ausencia de relaciones de naturaleza comercial o financiera que pudiesen constituir un conflicto de intereses.
Agradecemos el apoyo recibido del Ministerio de Ciencia e Innovación de España a través del programa de Apoyo a Centros de Excelencia Severo Ochoa 2019-2023 (CEX2018 000806S) y de la Generalitat de Catalunya a través de la Institució CERCA.
Este trabajo fue presentado en el XIII Congreso de la Sociedad Española de Reumatología Pediátrica (SERPE) (Madrid, España; noviembre del 2019) y el Congreso Euro-KiDs 2021 (encuentro en línea; enero del 2021).