Las infecciones por rotavirus son la causa más frecuente de gastroenteritis aguda en la infancia. Existen disponibles vacunas para su prevención, pero su uso todavía es limitado y estos virus continúan infectando a la población infantil, principalmente en los meses fríos del año.
ObjetivosCaracterizar los genotipos G (VP7) y P (VP4) de rotavirus detectados en niños con gastroenteritis aguda y determinar la prevalencia de las cepas de rotavirus con genotipo G9 en 3 departamentos de Salud Pública de la Comunidad Valenciana.
Pacientes y métodosSe estudiaron de forma prospectiva 541 muestras de heces de niños con gastroenteritis desde octubre de 2005 a septiembre de 2008. Se analizó la presencia de rotavirus por métodos de ELISA o inmunocromatografía y se identificaron los genotipos G y P de las cepas de rotavirus mediante transcripción inversa y amplificación en cadena de la polimerasa.
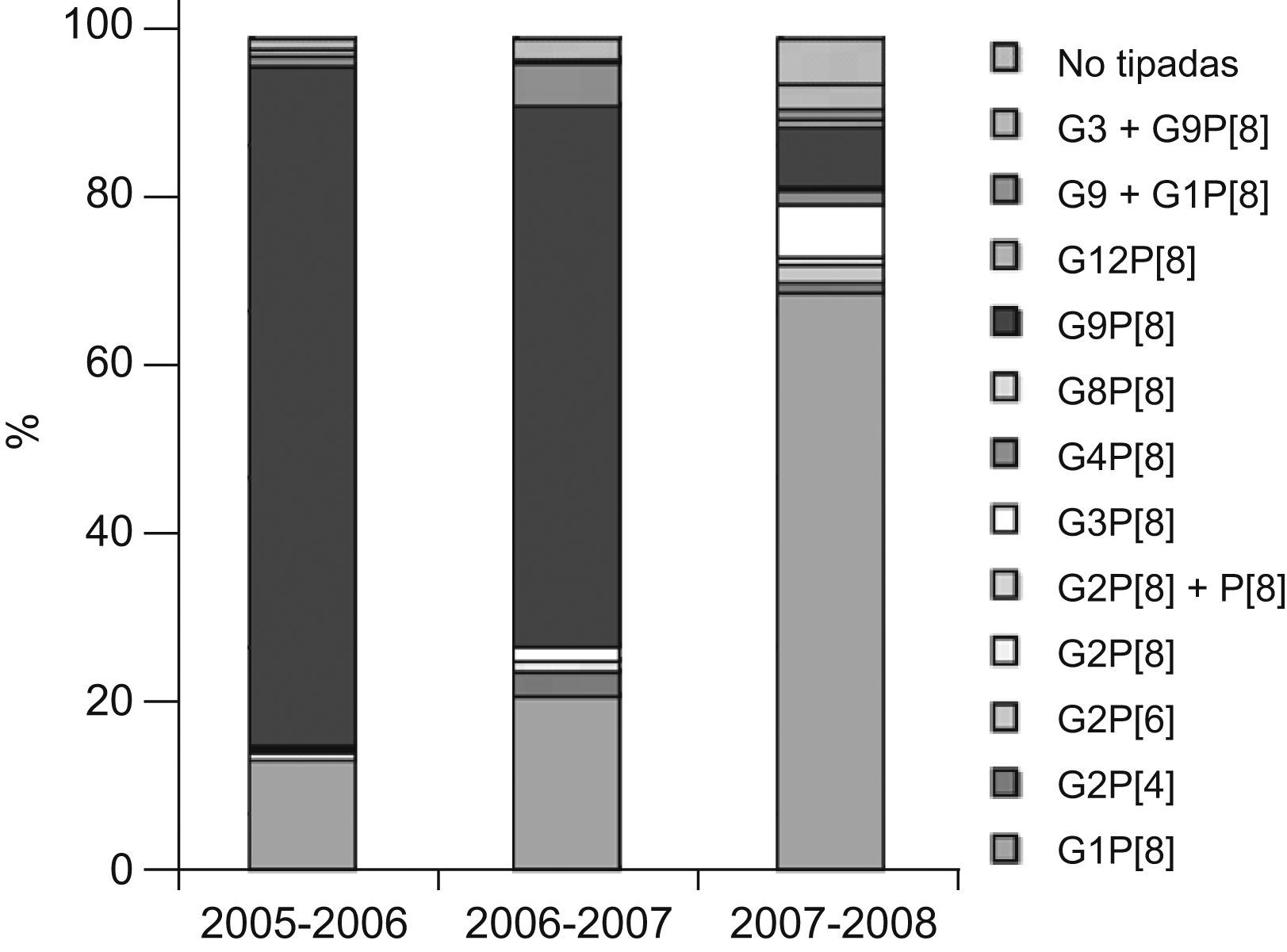
ResultadosSe caracterizaron los genotipos G y P en un total de 525 muestras (97%), y resultaron predominantes las cepas G9P[8] (56,5%) y G1P[8] (29,9%). Durante el período de tiempo estudiado, G9P[8] fue la combinación G/P más frecuente en las temporadas 2005–2006 y 2006–2007, y se encontró en el 81,2 y en el 64,7% de las muestras, respectivamente. Sin embargo, en la temporada 2007–2008 fueron las cepas G1P[8] las más frecuentes (68,8%), y G9P[8] disminuyó a un 7,2%.
ConclusionesLas cepas de rotavirus G9P[8] se diseminaron rápida y ampliamente durante los períodos 2005–2006 y 2006–2007, y suplantaron a otros genotipos anteriormente predominantes (G1, G4) en nuestra área geográfica. Su incidencia descendió bruscamente en la temporada 2007–2008, en la que las cepas G1P[8] resultaron ser nuevamente las que se detectaron más frecuentemente.
Rotavirus is the leading cause of acute gastroenteritis in young children worldwide. Effective vaccines to prevent rotavirus infections are currently available, although their clinical use is still limited, and rotavirus still causes many episodes of infantile gastroenteritis, mainly during the winter seasons.
ObjectiveTo characterise G (VP7) and P (VP4) genotypes of rotaviruses causing acute gastroenteritis in children and to determine the prevalence of genotype G9 rotavirus in three public health areas in the provinces of Valencia and Castellon.
Patients and methodsFive-hundred and forty-one stool samples were prospectively collected from infants and children with gastroenteritis in the period between October 2005 and September 2008. They were analysed for rotavirus by ELISA or by immunochromatography. G and P genotyping was performed by reverse transcription and PCR (RT-PCR).
ResultsG and P rotavirus genotypes were characterised in a total of 525 faecal samples (97%), resulting in a global predominance of strains G9P[8] (56.5%) and G1P[8] (29.9%). During the period of time studied, G9P[8] was the G/P combination most frequently detected during the rotavirus seasons 2005–2006 and 2006–2007, being present in 81.2% and 64.7% of the patients, respectively. However, during the 2007–2008 season, G1P[8] strains were the most frequently found (68.8%), with a sharp decrease in G9P[8] strains to 7.2% of the samples.
ConclusionsRotavirus G9P[8] have spread rapidly and widely during the 2005–2006 and 2006–2007 seasons, replacing other previously dominant genotypes (G1, G4) in our geographic area. Its incidence has declined sharply in 2007–2008, in which G1P[8] was again the predominating genotype.
La infección por rotavirus constituye la causa más frecuente de gastroenteritis aguda en la infancia, y es más prevalente en los niños menores de 5 años. Los rotavirus pertenecen a la familia Reoviridae y son virus sin envuelta, con una doble cápside que contiene un core central con el ARN genómico, compuesto por 11 segmentos de ARN de doble cadena. Los genes virales codifican proteínas estructurales y no estructurales; entre las primeras, destaca por sus características antigénicas la proteína VP6 de la cápside interna, que ha permitido clasificar los rotavirus en 7 serogrupos distintos (A-G) y es el grupo A el que infecta predominantemente a la especie humana. La glucoproteína VP7 de la cápside externa y la proteína VP4, que forma dímeros que constituyen las espículas del virión determinan la existencia de los serotipos/genotipos G y P, respectivamente. Los serotipos se establecen por ensayos de seroneutralización con anticuerpos policlonales o monoclonales y los genotipos G y P se identifican por análisis genómicos (hibridación o secuenciación). Hasta el momento, se han identificado 19 genotipos G y 27 genotipos P1.
Los genotipos G más comunes son G1, G2, G3 y G4 y los genotipos P más habituales son P[4] y P[8]. A mitad de la década de 1990 se detectó la aparición de un nuevo sero/genotipo G9 de rotavirus, con una amplia distribución geográfica2–5. El genotipo G9 se describió por vez primera en 1983 en Estados Unidos y se encontró con cierta frecuencia en la India y Tailandia en la década de 1980, pero desde 1995 ha resurgido y se ha diseminado por todos los continentes6. En la temporada invernal de 2001–2002 se lo encontró muy frecuentemente en varios países europeos (Italia, Bélgica, Suecia, Eslovenia)7–10. Este genotipo se ha descrito en combinación con P[4] y con P[8], pero también se ha encontrado asociado a P[6], P[9], P[11] y P[19]11. Actualmente existen disponibles vacunas frente a rotavirus del grupo A, monovalentes (G1P[8]) y pentavalentes (G1-G4, P1[8]), que parecen conferir protección cruzada frente a las infecciones por rotavirus G912,13.
Los objetivos de este estudio han sido caracterizar los genotipos G y P de rotavirus detectados en niños con gastroenteritis aguda en Valencia y Castellón, durante el período comprendido entre octubre de 2005 y septiembre de 2008, que incluye 3 temporadas epidémicas de rotavirus, y analizar la prevalencia de las cepas de rotavirus con genotipo G9 en estos períodos.
Pacientes y métodosSe recogieron de forma prospectiva las muestras de heces de niños menores de 5 años con gastroenteritis aguda atendidos en 3 hospitales de la Comunidad Valenciana: Hospital General de Castellón (Departamento de Salud 2), Hospital de Sagunto (Departamento de Salud 4) y Hospital Clínico Universitario de Valencia (Departamento de Salud 5), que ofrecen atención médica a una población de 285.309, 145.884 y 345.401 habitantes, respectivamente. Durante el período de estudio (de octubre de 2005 a septiembre de 2008) se analizaron 541 muestras de heces, de las que 207 correspondieron al Hospital Clínico Universitario de Valencia, 78 correspondieron al Hospital de Sagunto y 256 correspondieron al Hospital General de Castellón. Se excluyeron del estudio aquellas muestras de un mismo paciente recogidas con un intervalo inferior a 15 días, por considerar que correspondían a un mismo episodio diarreico, así como aquellas de las que se carecía de la información completa (nombre del paciente, edad, sexo, procedencia y fecha).
Estudio virológicoSe investigó la presencia de rotavirus en heces por el método de ELISA (Rotaclone®, Meridian Diagnostics, Cincinnati, Ohio, EE. UU.) o por inmunocromatografía (Vikia Rota-Adeno® de BioMérieux, Marcy l’Étoile, Francia, y Simple Rota-Adeno® de Operon, Cuarte, Zaragoza, España), y se descartó en todos los casos la presencia de bacterias enteropatógenas mediante la realización de coprocultivos en medios bacteriológicos. En las muestras que resultaron positivas para rotavirus se extrajo el ARN vírico por el método de fenol-cloroformo y purificación con celulosa CF1114.
Transcripción inversa y amplificación en cadena de la polimerasaCon cada muestra de ARN vírico se realizó una reacción de transcripción inversa con cebadores aleatorizados (random primers) (Invitrogen, Paisley, Reino Unido) y posterior amplificación con oligonucleótidos específicos del gen de la proteína VP715 y con oligonucleótidos específicos del gen de la proteína VP416.
Tipificación de los genes de VP7 (G) y de VP4 (P)Los genotipos G y P de las cepas de rotavirus se determinaron separadamente, mediante reacciones en cadena de la polimerasa (PCR) múltiple semianidada, y se utilizó un cebador común y una mezcla de cebadores específicos de cada genotipo G y P15–19, que generaban un amplímero de un tamaño característico para cada uno de ellos. A partir del ADN complementario obtenido en la primera reacción de PCR, se realizó una segunda PCR semianidada con oligonucleótidos específicos de cada genotipo, según las secuencias de las regiones hipervariables de cada gen. Posteriormente se analizaron los productos amplificados mediante electroforesis en gel de agarosa al 1,5% en tampón tris-borato-EDTA con bromuro de etidio (0,5μg/ml) durante 1h a 70 voltios, y se observaron con un transiluminador de luz ultravioleta y un captador de imágenes (Gelprinter Ultra®, TDI, Alcobendas, Madrid, España).
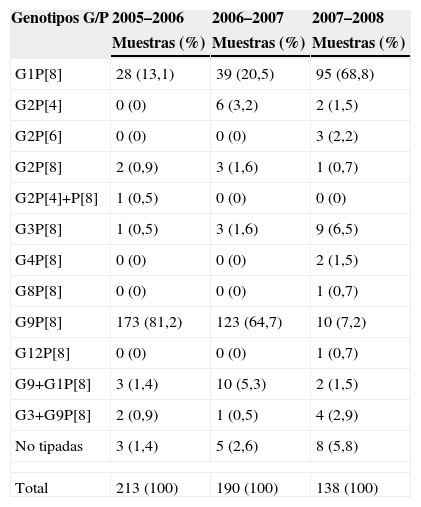
ResultadosDel total de 541 muestras de heces estudiadas, en las que previamente se había detectado la presencia de rotavirus por métodos inmunológicos, se han caracterizado los genotipos G y P de 525 cepas de rotavirus (97%) correspondientes a otros tantos pacientes, cuya distribución por sexos fue de 273 niños (52%) y 252 niñas (48%). La edad media fue de 1,6 años y los porcentajes de infección por grupos etarios fue la siguiente: el 41%, menores de 1 año; el 53,4%, de 1 a 4 años; el 2,7%, de 5 a 9 años, y el 2,9%, de 10 a 14 años. Dentro del grupo con mayor porcentaje de infección (1 a 4 años) se observó una mayor incidencia en los pacientes con edades comprendidas entre 12 y 24 meses (46%), seguidos por los de 2 a 3 años (28%) y por los de 3 a 4 años (26%). Los genotipos G y P detectados y su distribución por cada temporada de rotavirus (considerada de octubre a septiembre del año siguiente) se presentan en la tabla 1. Asimismo, en la figura 1 se representa gráficamente la distribución de los genotipos G/P en Valencia y Castellón en cada temporada estudiada.
Número de muestras y porcentajes de distribución de las combinaciones de genotipos G/P durante las temporadas de rotavirus 2005–2006, 2006–2007 y 2007–2008 en Valencia y Castellón
| Genotipos G/P | 2005–2006 | 2006–2007 | 2007–2008 |
| Muestras (%) | Muestras (%) | Muestras (%) | |
| G1P[8] | 28 (13,1) | 39 (20,5) | 95 (68,8) |
| G2P[4] | 0 (0) | 6 (3,2) | 2 (1,5) |
| G2P[6] | 0 (0) | 0 (0) | 3 (2,2) |
| G2P[8] | 2 (0,9) | 3 (1,6) | 1 (0,7) |
| G2P[4]+P[8] | 1 (0,5) | 0 (0) | 0 (0) |
| G3P[8] | 1 (0,5) | 3 (1,6) | 9 (6,5) |
| G4P[8] | 0 (0) | 0 (0) | 2 (1,5) |
| G8P[8] | 0 (0) | 0 (0) | 1 (0,7) |
| G9P[8] | 173 (81,2) | 123 (64,7) | 10 (7,2) |
| G12P[8] | 0 (0) | 0 (0) | 1 (0,7) |
| G9+G1P[8] | 3 (1,4) | 10 (5,3) | 2 (1,5) |
| G3+G9P[8] | 2 (0,9) | 1 (0,5) | 4 (2,9) |
| No tipadas | 3 (1,4) | 5 (2,6) | 8 (5,8) |
| Total | 213 (100) | 190 (100) | 138 (100) |
El genotipo G9P[8] fue claramente predominante durante las temporadas 2005–2006 y 2006–2007, y se encontró en 173 (81,2%) pacientes y 123 (64,7%) pacientes, respectivamente. En ambas temporadas, el segundo genotipo más frecuentemente detectado fue G1P[8] en 28 (13,1%) y 39 (20,5%) casos, respectivamente. Sin embargo, en la temporada 2007–2008 se observó una disminución muy importante del genotipo G9P[8], pues sólo se detectó en 10 muestras (7,2%), mientras que el genotipo G1P[8] fue el más prevalente, con 95 casos (68,8%). En esta temporada se detectaron también 2 cepas G4P[8] (1,5%) y sendas cepas G8P[8] (0,7%) y G12P[8] (0,7%).
Se observó la presencia de infección mixta en 23 muestras (4,3%), producidas por los genotipos G1+G9 en 15 muestras, G3+G9 en 7 muestras y G2P[4]+P[8] en una muestra.
DiscusiónLa identificación de los genotipos de rotavirus causantes de gastroenteritis en la población infantil es importante, ya que permite conocer su distribución en un área geográfica y en un período de tiempo determinados. Diferentes estudios han demostrado que en una misma comunidad pueden circular simultáneamente distintos genotipos, y que éstos pueden ir variando a lo largo del tiempo4,20,21. En este trabajo se ha analizado la epidemiología molecular de rotavirus en niños con diagnóstico de gastroenteritis aguda en 3 áreas de Salud Pública de la Comunidad Valenciana desde octubre de 2005 a septiembre de 2008.
En nuestro análisis de los casos con infección por rotavirus, el mayor porcentaje correspondió a varones con una edad comprendida entre 1 y 2 años, observación que coincide con estudios realizados en otras áreas22,23.
Se ha logrado caracterizar el genotipo G y P en 525 muestras de un total de 541 (97%). Las causas por las que no se logró tipificar 16 muestras pueden ser muy diversas, desde la presencia en ellas de inhibidores de la PCR, la degradación del ARN vírico, la existencia de mutaciones en las secuencias reconocidas por los cebadores hasta la existencia de genotipos no investigados. En ocasiones, esto puede deberse a resultados falsos positivos de los métodos inmunológicos empleados para establecer el diagnóstico, ELISA o inmunocromatografía, como se ha demostrado en nuestro laboratorio24.
En la mayoría de los estudios epidemiológicos de rotavirus se han determinado los tipos G por la importancia que han tenido estos serotipos en el diseño de las vacunas y por el valor que se ha atribuido a los anticuerpos neutralizantes anti-VP7 en conferir protección frente a las infecciones de rotavirus. En nuestro estudio hemos observado un claro predomino del genotipo G9P[8] seguido del G1P[8] durante las temporadas 2005–2006 y 2006–2007, con una menor presencia de cepas con genotipo G2, G3 y la ausencia de G4. Este último dato es interesante, ya que G4 fue uno de los genotipos más comunes en Valencia durante los años 1996–19994 y uno de los más comunes en España en la década de 199023,29. Sin embargo, en la temporada 2007–2008 se produce un brusco descenso del genotipo G9P[8], que pasa a constituir solamente el 7,2% de las cepas detectadas, y de nuevo G1P[8] es el más prevalente (68,8%). Es interesante destacar que en esta temporada se detectaron 2 genotipos muy poco frecuentes, identificados según nuestros datos por vez primera en España, G8P[8] y G12P[8], cada uno de ellos en un paciente.
El genotipo G9 de rotavirus se identificó por primera vez en 1983 en los Estados Unidos25 y durante el período 1985–1986 en Japón26,27. Posteriormente se describió en Yugoslavia en 1985–1988, en Tailandia en 1989 y en la India de 1986 a 199328. La aparición en diferentes áreas del mundo del genotipo G9 ha despertado un considerable interés, incluyendo sus aspectos epidemiológicos, su origen evolutivo y la composición genética del virus, así como su posible impacto en la eficacia de una vacuna frente a rotavirus12,13. En España se detectaron por vez primera cepas de rotavirus G9 en Madrid en 1998–199929. En nuestro laboratorio se identificaron las primeras cepas G9 en la temporada 2003–2004, y en 2004–2005 constituyeron el 10,7% de las cepas estudiadas (Abu-Mallouh, tesis doctoral, Universidad de Valencia, 2006). Como se demuestra en el presente estudio, en 2005–2006 se produjo una rápida diseminación de este genotipo, que se convirtió en el más prevalente en esa temporada y en la siguiente, disminuyendo drásticamente en 2007–2008. No se conocen los motivos de la distribución tan rápida de este genotipo en nuestra zona geográfica y de su implantación como genotipo predominante en 2 temporadas seguidas (2005–2006 y 2006–2007), hecho que también se ha descrito en otras áreas de la Península Ibérica, como Portugal30. Una posible explicación es que, al tratarse de una cepa vírica nueva, la población no presentaba inmunidad protectora frente a este serotipo, por lo que se produjo una rápida transmisión de éste. Las frecuentes infecciones por G9 habrían provocado el desarrollo en la población de inmunidad frente a este nuevo sero/genotipo, lo que produjo un brusco descenso de su incidencia en 2007–2008. Además, si las vacunas de rotavirus comercializadas son realmente protectoras frente a G9, a pesar de que no lo incluyen en su composición, podría suceder que las infecciones naturales con otros genotipos induzcan también protección cruzada frente a G9. Recientemente se ha publicado un análisis filogenético de 12 cepas de rotavirus G9 aisladas en Madrid, y todas ellas resultaron pertenecer al linaje III de los 6 linajes descritos31. En cuanto al genotipo P, el predominante ha sido P[8], seguido a distancia por P[4] y P[6], con resultados similares en otros estudios3,32,33.
Santos y Hoshino realizaron una revisión de 124 trabajos que incluyen un total de 45.571 cepas de rotavirus, en la que los 4 tipos G más comunes (G1–G4) en asociación con P[8] o P[4] representan más del 88% de las cepas halladas en todo el mundo, panorama que ha ido variando durante los años, fundamentalmente a causa de la aparición de G9 asociado con P[8] o con P[6]34.
Hemos detectado 23 casos de infecciones mixtas, en las que se encontraron más de un genotipo G o P. Este hallazgo es también frecuente en otros estudios: se encontró hasta un 31% de infecciones mixtas en un estudio realizado en la India35. Las nuevas combinaciones G/P detectadas en los últimos años podrían deberse al incremento de la inmigración procedente de otros países.
La introducción de las vacunas para prevenir las infecciones de rotavirus justifica el interés de este tipo de estudios para conocer los genotipos de rotavirus circulantes en las distintas áreas geográficas. Durante el período de tiempo estudiado en este trabajo se produjo la comercialización de las vacunas frente a rotavirus en España, sin que pueda atribuirse a este hecho el predominio del genotipo G9 durante estos últimos años. Es posible que las cepas víricas circulantes se vean condicionadas en el futuro por el impacto de un uso cada vez más amplio de las vacunas, ya que éstas podrían favorecer la aparición de variantes víricas resistentes a los anticuerpos neutralizantes que inducen. Sin embargo, la evolución de las cepas víricas se produce de forma espontánea, sin poderse atribuir los reemplazos y las emergencias de genotipos a una causa determinada. Por todo esto, el interés de los estudios de epidemiología molecular de rotavirus está hoy plenamente justificado.
FinanciaciónEuropean Rotavirus Network (EuroRotaNet). Red coordinada por el Dr. J. Gray, Health Protection Agency, Colindale, Reino Unido, y Rotavirus Vaccine Program, OMS; cofinanciada por GlaxoSmithKline Biologicals (GSK) y Sanofi Pasteur-MSD.
Este trabajo se presentó parcialmente como comunicación oral en el 56.o Congreso de la Asociación Española de Pediatría, celebrado en Barcelona, 7–9 junio de 2007.