Editado por: Fernando Santos-Simarro. Molecular Diagnostic and Clinical Genetics Unit University Hospital Son Espases. Palma de Mallorca. España
Última actualización: Septiembre 2025
Más datosEl desarrollo de tecnologías genómicas ha transformado la práctica de la pediatría, permitiendo avances significativos en el diagnóstico de enfermedades genéticas, el 70% de las cuales comienzan en la infancia. Las variaciones genómicas, que van desde cambios en un solo nucleótido hasta grandes reorganizaciones cromosómicas, son responsables de muchas enfermedades pediátricas, y su detección depende de la selección adecuada de tecnologías. Métodos como el cariotipo, MLPA, microarrays, secuenciación Sanger y secuenciación de nueva generación (Next Generation Sequencing [NGS]) han incrementado la capacidad diagnóstica, aunque, en general, solo el 27% de los casos pediátricos alcanzan un diagnóstico definitivo. Los paneles de genes, exomas, genomas y RNAseq ofrecen diferentes rendimientos diagnósticos según la complejidad del caso clínico, con tasas que pueden alcanzar hasta el 75% en cohortes específicas. Además, tecnologías emergentes como la secuenciación de lectura larga y el mapeo óptico del genoma han demostrado utilidad en la identificación de variantes estructurales complejas y regiones repetitivas del genoma. La integración de un fenotipado clínico exhaustivo y herramientas como el vocabulario estandarizado de fenotipos de Human Phenotype Ontology (HPO) optimiza la priorización de variantes genéticas y mejora la precisión diagnóstica. Este artículo revisa las capacidades, las limitaciones y las aplicaciones clínicas de las técnicas genómicas disponibles en la actualidad, resaltando diferencias, ventajas y desventajas, y sus implicaciones para el diagnóstico en pediatría.
The development of genomic technologies has transformed pediatric practice, enabling significant advances in the diagnosis of genetic diseases, 70% of which manifest during childhood. Genomic variants, ranging from single-nucleotide changes to large chromosomal rearrangements, are responsible for many pediatric conditions, and their detection relies on the appropriate selection of technologies. Methods such as karyotyping, MLPA, microarrays, Sanger sequencing, and next-generation sequencing (NGS) have increased diagnostic capacity, although, on average, a definitive diagnosis is currently made in only 27% of pediatric cases. Gene panels and exome, genome, and RNA sequencing offer varying diagnostic yields depending on clinical complexity, with rates that may be as high as 75% in specific cohorts. Additionally, emerging technologies such as long-read sequencing and optical genome mapping have proven useful in identifying complex structural variants and repetitive genomic regions. The integration of comprehensive clinical phenotyping and tools like the Human Phenotype Ontology (HPO) standard vocabulary optimizes genetic variant prioritization and enhances diagnostic accuracy. This article reviews the capabilities, limitations and clinical applications of currently available genomic techniques, highlighting their differences, advantages and disadvantages as well as implications for diagnostics in pediatrics.
El avance en tecnologías genómicas ha revolucionado el diagnóstico de enfermedades minoritarias de origen genético, el 70% de las cuales se manifiestan en la infancia1. El desarrollo de herramientas de análisis eficientes han permitido incorporar la secuenciación de nueva o segunda generación (NGS) y los microarrays en la práctica clínica. Aunque estas técnicas han mejorado la capacidad diagnóstica, solo el 26,7% de los casos pediátricos logran un diagnóstico definitivo2.
El proceso de diagnóstico puede ser largo, complejo y costoso. Actualmente el tiempo promedio estimado para un diagnóstico preciso es de 5 años y alrededor del 30% de los niños con una enfermedad minoritaria mueren antes de los 5 años1. Durante este tiempo, tanto los pacientes como sus familias enfrentan desafíos emocionales y económicos significativos, mientras que el sistema de salud soporta una gran presión.
El análisis de la información genómica se ha convertido hoy en día en una herramienta imprescindible en el ámbito clínico. Contar con un diagnóstico genético contribuye a:
- •
Comprender la causa, la progresión de la enfermedad y la esperanza de vida.
- •
La identificación de otros familiares que puedan estar en riesgo de padecer la misma afectación, y ayudar con la planificación familiar mediante un adecuado asesoramiento genético.
- •
Poder acceder a tratamientos específicos (en el caso que existan para esa enfermedad) o participar en ensayos clínicos para el desarrollo de un tratamiento.
- •
Poder contactar con otras familias en la misma situación, y compartir experiencias y recursos.
Para maximizar la utilidad de las técnicas actuales de biología molecular, es esencial un fenotipado exhaustivo y preciso del paciente, que incluya una recopilación detallada de síntomas, manifestaciones clínicas y antecedentes médicos. Un fenotipado adecuado mejora el análisis de los datos genéticos, optimiza tiempos y reduce el riesgo de resultados ambiguos o no concluyentes.
La NGS puede detectar miles de variantes genéticas en un paciente, muchas de las cuales son de significado incierto (VUS). Sin embargo, un fenotipo claro permite priorizar las variantes más relevantes para las manifestaciones clínicas del paciente, descartando las menos significativas. Asimismo, los análisis del caso índice junto a sus padres (análisis en trío) permiten evitar en muchos casos variantes de significado incierto, priorizando variantes que son de novo o aquellas recesivas compuestas, heredadas de ambos progenitores.
Una herramienta ampliamente utilizada para estandarizar fenotipos y relacionarlos con genes, es la Human Phenotype Ontology (HPO) https://hpo.jax.org/3. El uso de HPO permite vincular datos fenotípicos directamente con datos genéticos, agilizando el análisis y priorización de variantes relevantes.
Tipos de variación en el genoma y su relación con la enfermedadEn el ámbito de la genómica, las variaciones en nuestro ADN son la base de la diversidad humana y, en muchos casos, la causa de enfermedades. Estas variaciones pueden ser de diferentes tipos y su tamaño y localización en el genoma pueden tener distintos efectos sobre la salud. Las Structural Variants (SVs) o variaciones estructurales, son grandes alteraciones en la estructura de los cromosomas, como deleciones, duplicaciones, inversiones o translocaciones. Estas variaciones pueden afectar a grandes regiones del genoma y, por lo tanto, a múltiples genes. Las Copy Number Variations (CNVs) o variaciones en el número de copias son un tipo específico de SV que implica un aumento o disminución en el número de copias de una región particular del genoma. Ejemplos de enfermedades causadas por CNVs son el síndrome de Williams-Beuren causado por una deleción en el cromosoma 7q11.23, el síndrome del maullido del gato causado por una deleción en el brazo corto del cromosoma 5p15.2, o el síndrome de Prader-Willi y Angelman causados por deleciones en el cromosoma 15q11.2-q13. Las Single Nucleotide Variant (SNVs) o variantes de un solo nucleótido son cambios en un nucleótido. Aunque son cambios pequeños, pueden tener un gran impacto, sobre todo si ocurren en regiones codificantes de proteínas o zonas reguladoras de la expresión génica. Algunos ejemplos de enfermedades habitualmente causadas por este tipo de variación son la fibrosis quística, que está causada por mutaciones en el gen CFTR, la anemia falciforme, causada por una mutación en el gen de la hemoglobina, o la fenilcetonuria, causada por mutaciones en el gen PAH. Finalmente, un tercer tipo de variación en el genoma son las Short Tandem Repeats (STRs) o repeticiones cortas en tándem, que son secuencias cortas de ADN que se repiten en tándem un número variable de veces y que pueden expandirse hasta niveles patológicos. La mayor parte de enfermedades causadas por una expansión de STRs afectan al sistema neurológico. Algunos ejemplos, serían el síndrome de X-frágil, que está causado por la expansión de la secuencia CGG en el gen FMR1 o la distrofia miotónica, que está causada por una expansión del triplete CTG en el gen DMPK. En resumen, las SVs, CNVs, SNVs y las expansiones de STRs son diferentes tipos de variaciones genéticas que pueden causar enfermedades. La identificación de variantes es fundamental para el diagnóstico, el asesoramiento genético y el desarrollo de nuevas terapias. Sin embargo, cada una de las técnicas diagnósticas disponibles no necesariamente detecta todos los tipos de variantes. Por todo ello y debido a los rápidos avances tecnológicos, es común dudar a la hora de solicitar un estudio genético, surgiendo preguntas como: ¿qué técnicas y pruebas ofrecen un mejor rendimiento hoy en día atendiendo al fenotipo del paciente? ¿Cuáles son las ventajas y las limitaciones de cada una de ellas? ¿Es mejor una prueba dirigida a un gen o síndrome concreto o pruebas no dirigidas que abarcan el genoma entero? A continuación, se intentará dar respuesta a estas preguntas.
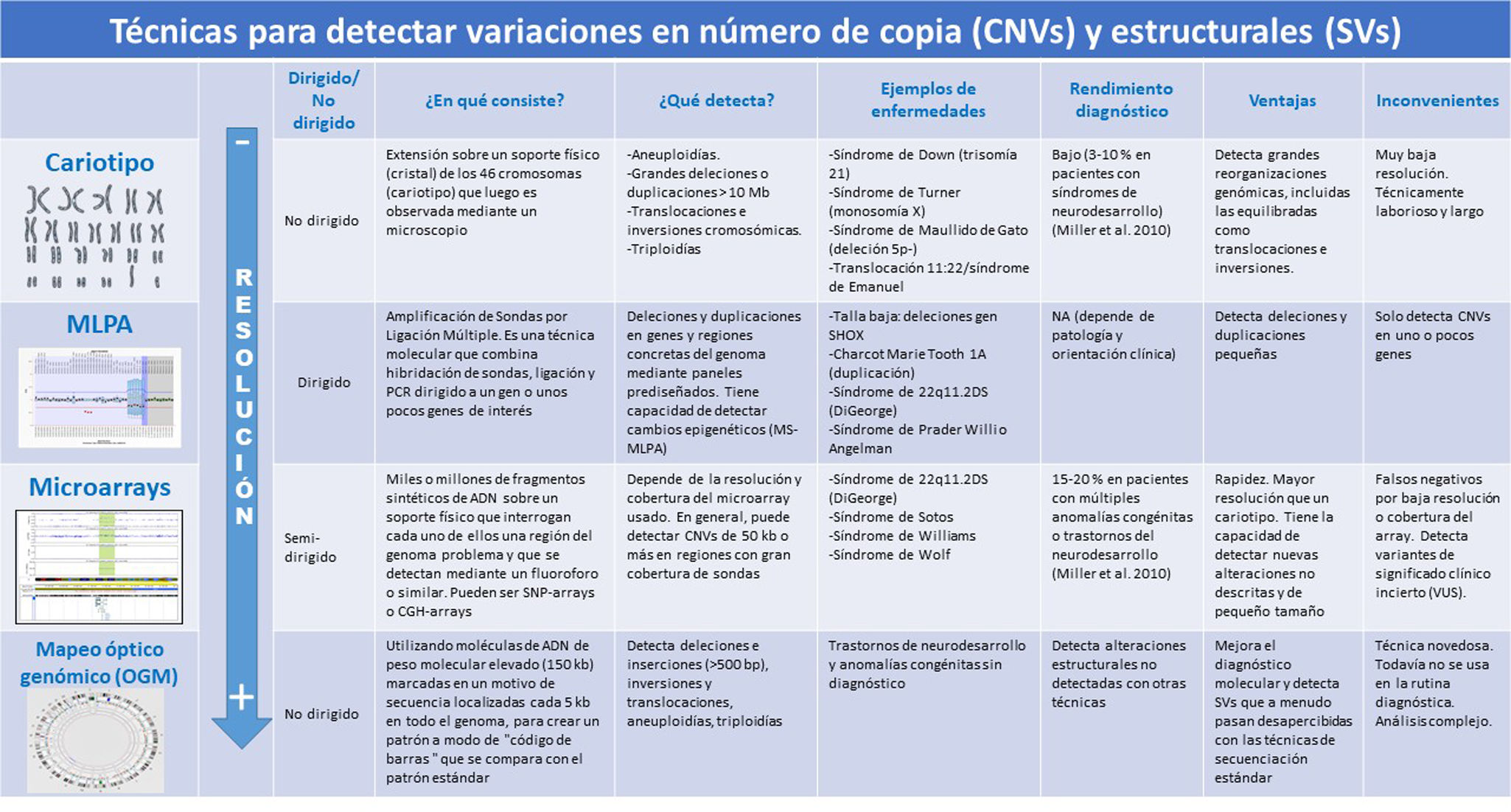
Comparación de cariotipo, MLPA y microarrays para la detección de SVs y CNVsLa comparación entre cariotipo, Multiplex Ligation-dependent Probe Amplification (MLPA) y microarrays para la detección de SVs en enfermedades genéticas pediátricas revela que cada técnica tiene fortalezas y limitaciones, lo que determina su utilidad en función del contexto clínico y diagnóstico (fig. 1).
Comparación de las diferentes técnicas que permiten detectar las CNVs y las SVs.
Fuente: Miller et al. 20104.
El cariotipo ha sido durante décadas la herramienta principal para analizar el número y la estructura de los cromosomas. Éste permite identificar grandes alteraciones cromosómicas, como aneuploidías (cambios en el número de cromosomas, como en el síndrome de Down, caracterizado por una trisomía 21) y reordenamientos estructurales evidentes, como translocaciones, inversiones o grandes deleciones. Sin embargo, su resolución es limitada, ya que solo puede detectar alteraciones que abarcan más de 5-10Mb (millones de bases). Esta baja sensibilidad impide que el cariotipo sea útil para identificar microdeleciones o microduplicaciones que son la causa de los síndromes microdelecionales involucrados en muchas enfermedades genéticas pediátricas. A pesar de ello, sigue siendo una técnica fundamental para ciertos contextos clínicos, como el diagnóstico de aneuploidías o translocaciones equilibradas.
El MLPA representa un avance significativo en la capacidad de detección de variaciones genéticas específicas. Esta técnica molecular utiliza sondas diseñadas para regiones particulares del genoma y permite identificar duplicaciones o deleciones en genes de interés. También existe una modalidad de MLPA (MS-MLPA) que detecta cambios en la metilación del genoma asociados a síndromes de la impronta. Es especialmente útil cuando se sospecha de síndromes genéticos concretos, como la distrofia muscular de Duchenne, causada por alteraciones en el gen DMD (MLPA), o los síndromes de Prader-Willi y Angelman (MS-MLPA). La alta sensibilidad del MLPA en estos casos contrasta con su limitación de no poder detectar variaciones estructurales complejas (como inversiones o translocaciones) ni alteraciones fuera de las regiones específicas analizadas por las sondas. Por esta razón, su uso se restringe a situaciones en las que la sospecha clínica esté claramente dirigida.
En contraste, los microarrays representan una herramienta poderosa para el análisis genómico a gran escala, permitiendo la detección de CNVs a partir de aproximadamente 50kb. Su cobertura del genoma es significativamente mayor que la del cariotipo o la MLPA, lo que los hace especialmente útiles en casos sin una sospecha clínica específica. Esta tecnología facilita la identificación de microdeleciones y microduplicaciones en todo el genoma, contribuyendo al diagnóstico de trastornos genéticos que podrían pasar desapercibidos con métodos convencionales. Si bien la mayoría de los microarrays no están diseñados para detectar mutaciones puntuales ni algunas variaciones estructurales complejas, ciertos formatos avanzados (SNP-arrays) pueden identificar SNVs y regiones de pérdida de heterocigosidad. Aun así, en diagnóstico, su principal utilidad radica en la detección de alteraciones cromosómicas submicroscópicas, lo que los convierte en una herramienta de primera línea en el estudio de enfermedades genéticas. Ejemplos notables incluyen la detección de CNVs subteloméricas como causa de discapacidad intelectual idiopática o las duplicaciones en el cromosoma 16p11.2, vinculadas al autismo.
Dado que cada técnica tiene sus propias aplicaciones y limitaciones, en la práctica clínica, a menudo se combinan varias de estas herramientas para un diagnóstico más completo, dependiendo de los fenotipos y las sospechas clínicas.
Comparación de la secuenciación Sanger y NGS y su aplicación en la detección de SNVs y SVsLa secuenciación de Sanger y la secuenciación NGS son 2 tecnologías utilizadas para analizar principalmente SNVs, pero tienen diferencias significativas en cuanto a capacidad, eficiencia, coste y tipos de variantes que pueden detectar (fig. 2).
La secuenciación de Sanger es una técnica que se utiliza principalmente para secuenciar fragmentos de ADN pequeños. Aunque es una tecnología más antigua, sigue siendo ampliamente utilizada en la práctica clínica sobre todo para estudios familiares de variantes ya conocidas y que segregan con la enfermedad. Tiene una alta resolución para secuenciar genes específicos o exones, y aunque está limitada a secuencias cortas (aproximadamente 600-800 bases), presenta una tasa de error extremadamente baja. Es ideal para mutaciones puntuales específicas, como las que se encuentran en un solo gen o una región pequeña del genoma. Es una opción válida para confirmación de diagnóstico cuando se sospecha de una mutación conocida en un gen específico.
La NGS, también conocida como de segunda generación o secuenciación masiva paralela, ha revolucionado el diagnóstico genético permitiendo una secuenciación a gran escala y a un coste proporcionalmente menor que las técnicas tradicionales. La NGS permite secuenciar grandes cantidades de ADN simultáneamente. Tiene capacidad para secuenciar paneles de genes, exomas completos (solo los exones de los genes) o genomas completos con alta resolución. Detecta SNVs, pero también CNVs y SVs. Aunque la NGS es más costosa que el Sanger para análisis de pocos genes, presenta mayor eficiencia cuando se necesitan analizar grandes paneles de genes o el exoma completo. Puede reducir el tiempo total de diagnóstico en comparación con las pruebas convencionales, especialmente cuando hay múltiples genes implicados. En general, tiene una precisión comparable al Sanger, aunque las tasas de error pueden ser mayores, especialmente en regiones repetitivas. La NGS es especialmente útil en situaciones donde los fenotipos no son específicos o cuando se sospecha entre múltiples trastornos genéticos. Ejemplos incluyen: autismo, cardiopatías congénitas o discapacidad intelectual. Requiere de bioinformática avanzada para procesar, analizar e interpretar los datos, lo que puede ser una barrera. La identificación de variantes de significado incierto puede dificultar el establecimiento de un diagnóstico definitivo.
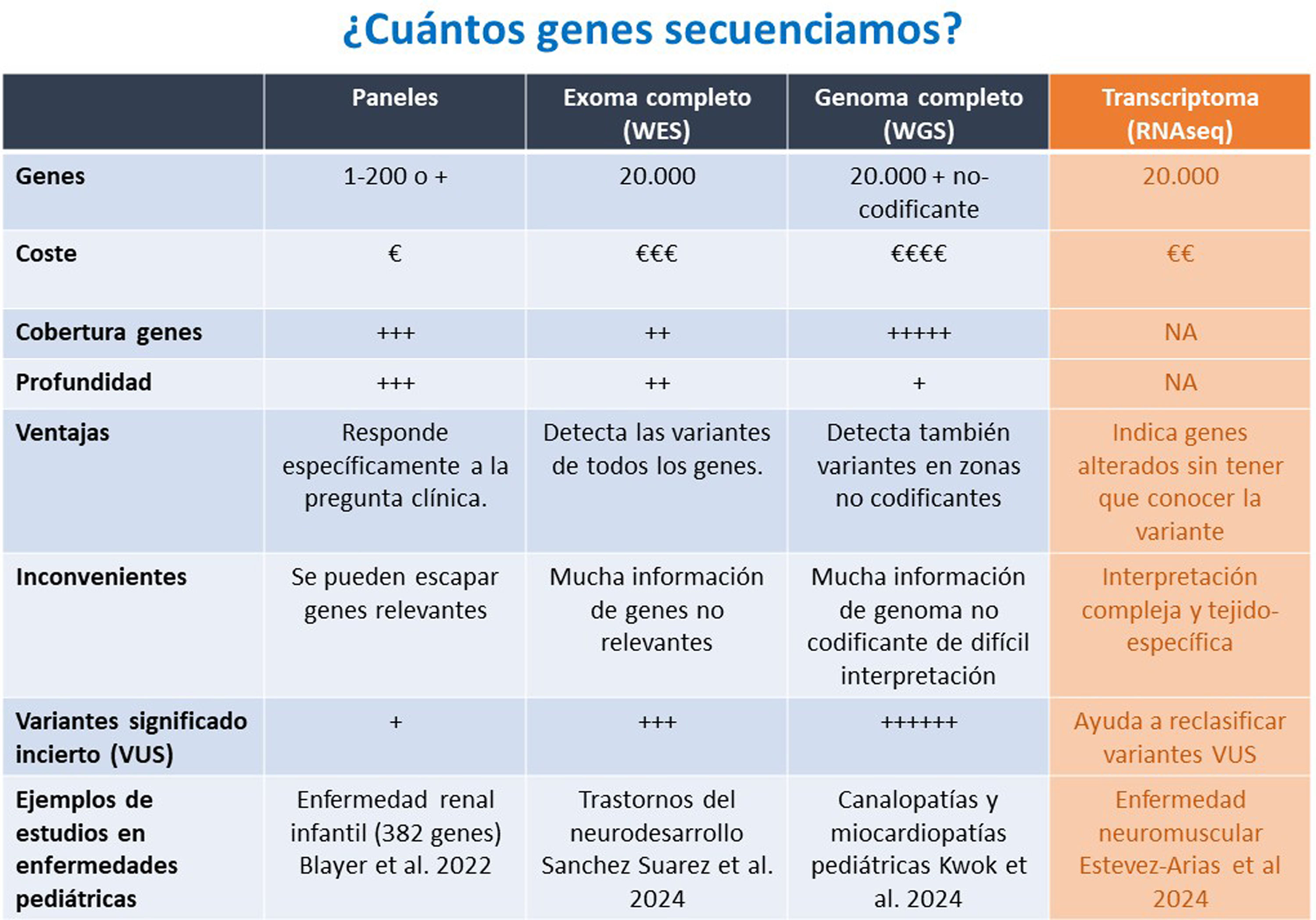
La utilidad de paneles, exomas, genomas y RNAseqLa utilidad de los paneles de genes, exomas, genomas y RNAseq en la secuenciación NGS, para el diagnóstico de enfermedades pediátricas varía según el contexto clínico, el presupuesto y los objetivos del análisis (fig. 3).
Los paneles de genes están dirigidos a un conjunto limitado de genes relacionados con enfermedades específicas. En general, se diseñan para cubrir exhaustivamente genes clave asociados con un fenotipo clínico concreto. Son más económicos en comparación con el exoma o el genoma completo y contienen menos datos que interpretar, lo que facilita una rápida identificación de variantes relevantes. Además, minimizan la posibilidad de identificar variantes genéticas no relacionadas con el problema clínico. Su principal desventaja es que puede fallar en el diagnóstico cuando el fenotipo está asociado a otros genes no contemplados en el panel. Son especialmente útiles para enfermedades monogénicas con sospechas diagnósticas claras (p. ej., distrofias musculares, Rendu-Osler, Alport).
El exoma (WES, del inglés Whole exome sequencing) es la parte del genoma que contiene los exones, que son las regiones codificantes de los genes responsables de producir proteínas. Al secuenciar el exoma, se analiza aproximadamente el 1-2% del genoma, pero sobre este pequeño porcentaje recaen hasta el 85% de las variantes conocidas relacionadas con enfermedades. Una ventaja sobre los paneles es que detecta variantes en genes inesperados que pueden explicar fenotipos complejos. Sin embargo, no detecta variantes en regiones reguladoras o intrónicas, que también pueden ser clínicamente relevantes. Además, se identifican muchas variantes, lo que puede aumentar el tiempo de interpretación respecto a un panel, así como un mayor riesgo de identificar variantes no relacionadas clínicamente. Está recomendado cuando el fenotipo es complejo o no se sospecha de un grupo específico de genes, aunque hoy en día se pueden crear paneles virtuales bioinformaticamente que eliminan los genes no relevantes para una enfermedad o fenotipo permitiendo el uso del exoma como si fueran paneles de genes.
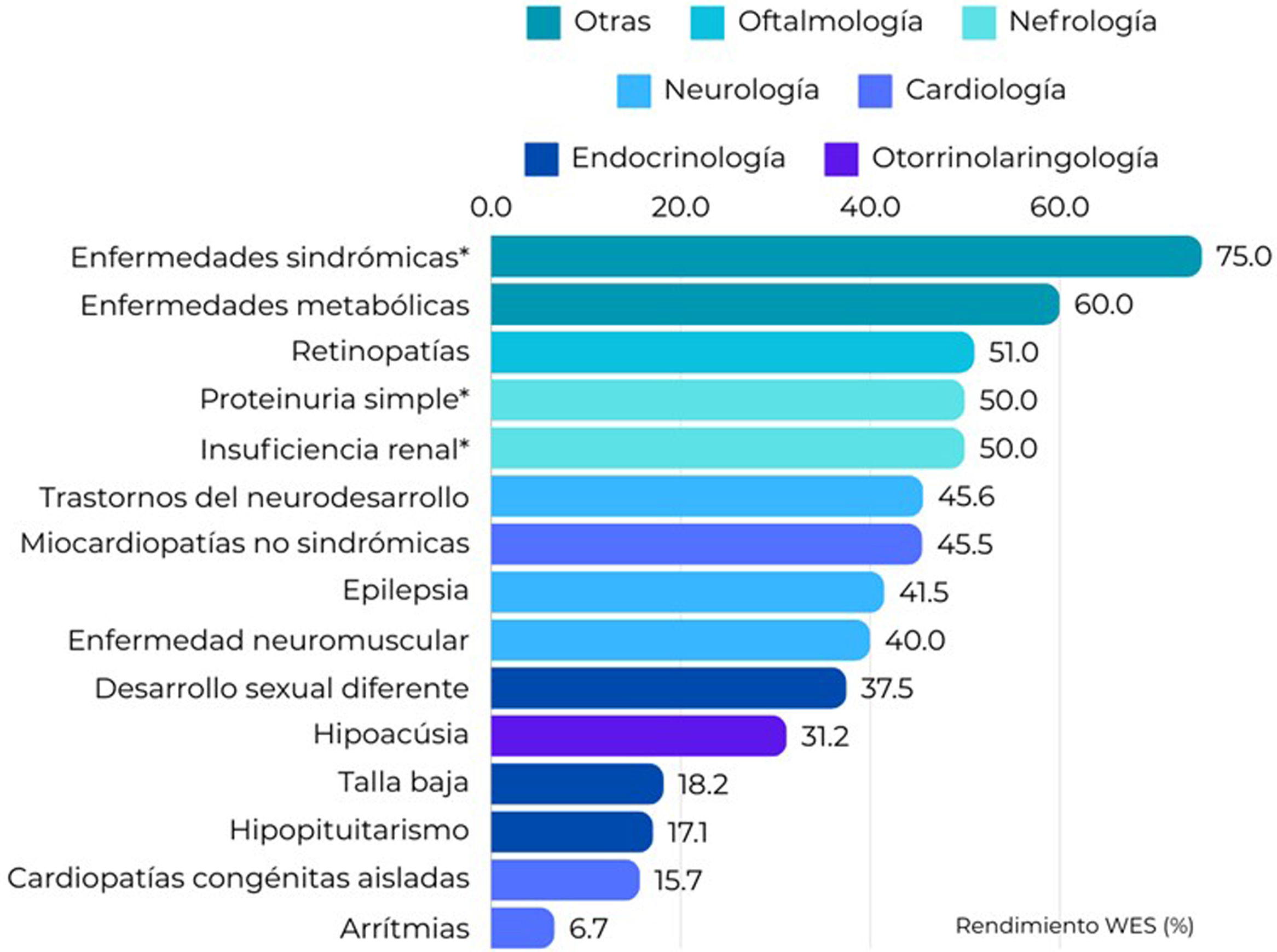
El rendimiento diagnóstico del WES aplicado a la práctica clínica varía entre el 10% o más del 60%, dependiendo de las características clínicas de la cohorte analizada, el año de la prueba y la estrategia analítica10. En la figura 4 se puede observar el rendimiento diagnóstico del WES calculado en diferentes estudios realizados en población pediátrica y publicados recientemente (2023-2024). Destaca el elevado rendimiento observado en los pacientes sindrómicos (trastornos del neurodesarrollo más otros fenotipos como dismorfia facial, epilepsia, cardiopatía congénita o distonía) cuando se realiza la secuenciación en trio que asciende hasta el 75%11. Por el contrario, los rendimientos más bajos se observan en arritmias y cardiopatías congénitas aisladas12.
Rendimiento diagnóstico del WES según el fenotipo estudiado calculado en las siguientes publicaciones científicas: 11–20. *Estudios en TRIO.
El genoma completo (WGS) es la secuenciación de toda la secuencia del ADN conocida. Es una prueba que abarca la mayoría de los tipos de variación al detectar variantes en regiones no codificantes, intrones y variantes estructurales, como inversiones, duplicaciones y deleciones (CNVs). Analizar el genoma completo puede evitar realizar otras pruebas específicas para detectar CNVs y SVs, como los microarrays, y podría ser una prueba de primera opción. Sin embargo, a día de hoy, no se encuentra todavía implantada en la rutina diagnóstica de la mayoría de laboratorios y se usa para enfermedades con etiología genética compleja o no conocida o que no se ha hallado variantes con otras técnicas o aproximaciones21. También, es adecuado para investigar nuevas asociaciones genéticas. Presenta un coste mayor que otros enfoques, aunque actualmente está disminuyendo. También requiere de mayor tiempo y recursos para su análisis e interpretación. Igual que el exoma, puede revelar información inesperada o no deseada (p. ej., predisposición a enfermedades adultas).
Agregar la secuenciación de RNA (RNAseq) a la comparación entre paneles de genes, exomas y genomas proporciona una perspectiva más amplia, ya que el RNAseq permite explorar la expresión génica y eventos relacionados con el procesamiento del RNA (splicing). El RNAseq secuencia el transcriptoma, abarcando el RNA mensajero y otras formas de RNA. Detecta cambios en la expresión génica, aberraciones en el splicing y transcritos de fusión. Es útil en el diagnóstico de enfermedades causadas por variantes que alteran la expresión o el procesamiento del RNA e identifica los efectos funcionales de variantes en regiones reguladoras no codificantes o intrónicas. Complementa el exoma y el genoma para validar hallazgos genéticos. Sin embargo, depende del tejido utilizado, y como principio general, el RNA debe provenir del tejido afectado para detectar alteraciones relevantes. Además, no detecta variantes genómicas si éstas no afectan al RNA. A día de hoy su uso no es habitual en el diagnóstico de rutina y se ciñe más al ámbito de la investigación.
La selección de la técnica depende del caso clínico. Un enfoque escalonado es común, comenzando con paneles o exomas, seguido de genoma completo o RNAseq si no se encuentra un diagnóstico claro. En enfermedades pediátricas complejas, la combinación de técnicas (p. ej., genoma+RNAseq) puede ser clave para maximizar la precisión diagnóstica. De cualquier manera, tanto WGS como RNAseq no son todavía de uso habitual en el diagnóstico en nuestro entorno y su uso se ciñe más al ámbito de la investigación.
Tecnologías genómicas emergentesLa secuenciación de lectura larga (TGS, del inglés Third Generation Sequencing) y el mapeo óptico del genoma (OGM, del inglés Optical Genome Mapping) son tecnologías avanzadas que complementan e incluso pueden sustituir herramientas como la NGS y los microarrays en ciertos contextos. Estas técnicas han demostrado ser especialmente útiles en el diagnóstico de enfermedades pediátricas, donde las variantes estructurales complejas, difíciles de detectar con NGS, pueden ser responsables de la enfermedad.
El OGM permite la detección de variantes estructurales en todo el genoma mediante la visualización física de moléculas de ADN extremadamente largas (hasta cientos de kilobases). Su alta sensibilidad lo hace ideal para identificar translocaciones, inversiones, grandes deleciones, duplicaciones y variantes en el número de copias (CNVs), incluidas aquellas en regiones repetitivas y no codificantes. A diferencia de la NGS, no requiere amplificación ni secuenciación, lo que reduce sesgos metodológicos. No obstante, presenta limitaciones en la detección de variantes pequeñas (< 500 pb) y su implementación sigue siendo costosa en términos de instrumentación y análisis. Su utilidad radica en la identificación de alteraciones estructurales no detectadas por NGS, como en algunos síndromes de deleción/duplicación o en ciertos casos de cáncer pediátrico.
Por otro lado, la secuenciación de lectura larga (TGS) genera fragmentos de ADN de gran longitud (kilobases), permitiendo un mapeo más preciso de regiones genómicas complejas. Es especialmente útil para detectar variantes estructurales difíciles de resolver con las lecturas cortas de la NGS y para analizar repeticiones en tándem, relevantes en enfermedades como la distrofia miotónica o el síndrome de X frágil. Además, cuando se aplica en RNAseq, facilita la identificación de isoformas alternativas del ARN. A pesar de sus ventajas, la TGS sigue siendo más costosa que la NGS y presenta una mayor tasa de error inicial, lo que requiere infraestructura especializada tanto para la generación de datos como para su análisis.
En conjunto, tanto el OGM como la TGS representan avances significativos para mejorar el diagnóstico en casos genéticos complejos, especialmente cuando las pruebas estándar (paneles, exoma o genoma) no han sido concluyentes. Con el tiempo, su evolución tecnológica y la reducción de costos determinarán si pueden reemplazar a la NGS como prueba diagnóstica de primera línea. Sin embargo, el aumento en el rendimiento de estas técnicas, sigue siendo limitado, con mejoras de no más del 10%, posiblemente debido a factores no genéticos o poligénicos.
ConclusionesLas nuevas tecnologías genómicas están revolucionando el diagnóstico en pediatría y mejorando la identificación de enfermedades genéticas, aunque aún existen desafíos en la interpretación y accesibilidad de estas pruebas. A medida que avanza la tecnología, la secuenciación del exoma y el genoma completo están cobrando mayor relevancia en la práctica clínica, facilitando diagnósticos más rápidos y precisos. Sin embargo, su implementación requiere de mejoras en bioinformática, interpretación de datos y accesibilidad. En este contexto, el fenotipado preciso es clave para optimizar el análisis genético y reducir la incertidumbre en los resultados. Además, la combinación de diferentes técnicas (cariotipo, MLPA, microarrays, NGS) permite una evaluación más exhaustiva de las variantes genómicas.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.