Cada año se notifican ciento treinta millones de infecciones por Chlamydia trachomatis en todo el mundo. Diecinueve serotipos de este patógeno pueden causar infecciones en mujeres embarazadas y recién nacidos. En México se desconoce la distribución de estos genotipos en recién nacidos con infecciones respiratorias.
Material y métodosSe analizaron mil sesenta y dos muestras de lavado bronquial de neonatos con síndrome de dificultad respiratoria para detección de infección por clamidia. El diagnóstico de clamidia se realizó mediante la detección de plásmidos con un ensayo PCR interno y los genotipos se identificaron mediante un ensayo PCR-RFLP del gen ompA.
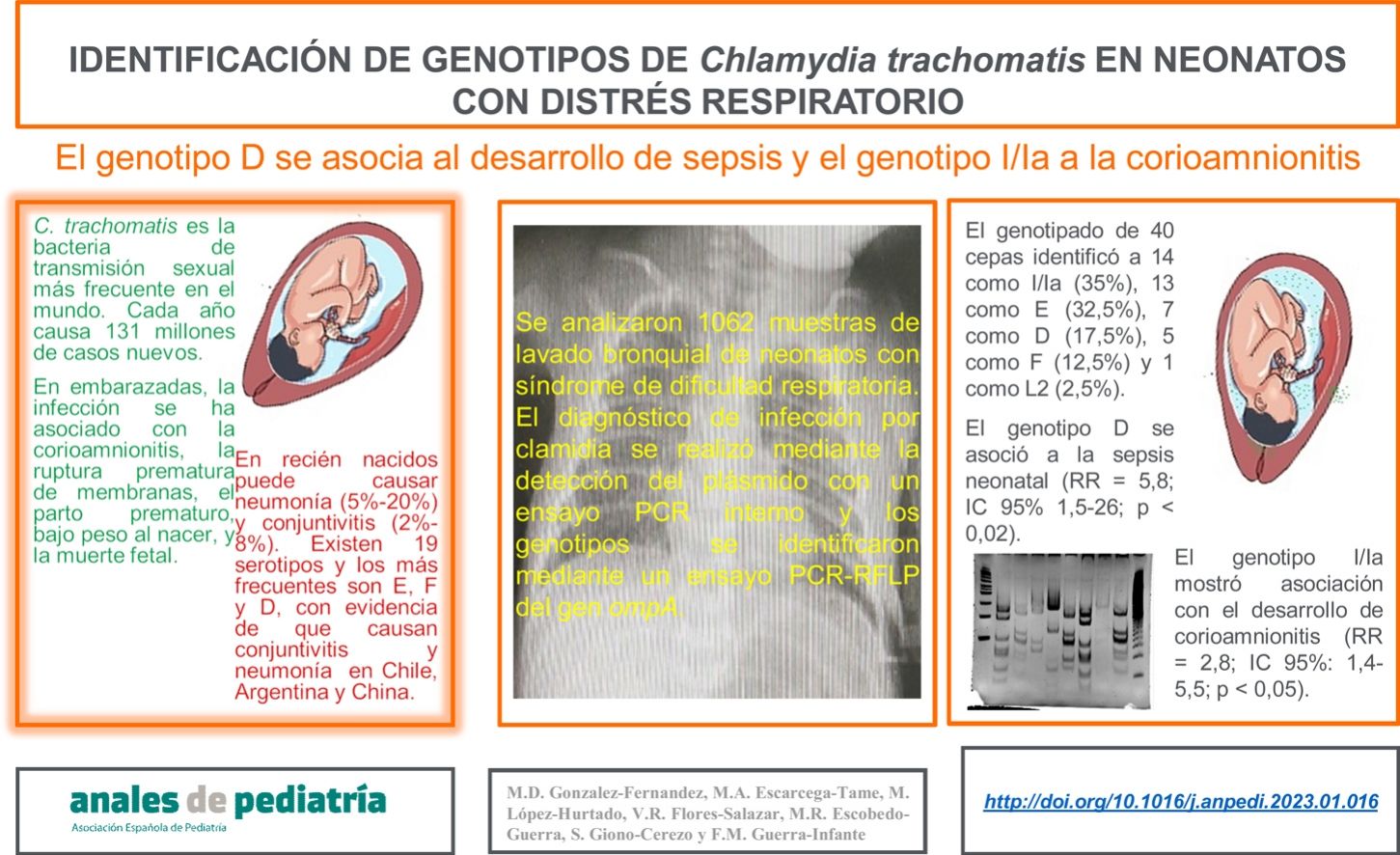
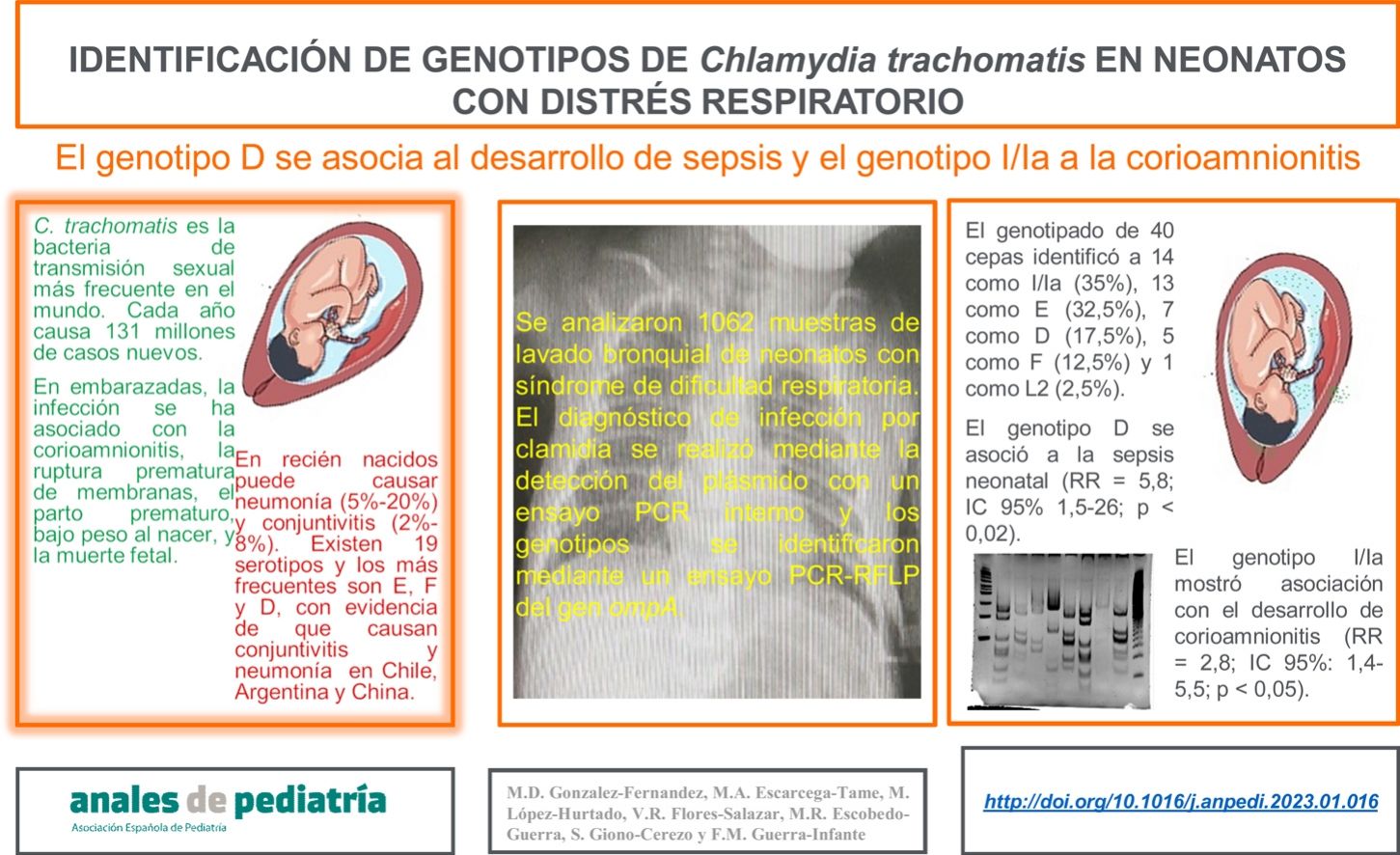
ResultadosEl genotipado de 40 cepas identificó a 14 como I/Ia (35%), 13 como E (32,5%), 7 como D (17,5%), 5 como F (12,5%) y 1 como L2 (2,5%). El análisis de riesgo relativo mostró que el genotipo D se asoció con sepsis neonatal (RR=5,83; IC 95%: 1,51-25,985; p <0,02), mientras que el genotipo I/Ia mostró asociación significativa con madres que desarrollaron corioamnionitis (2,8; IC 95%: 1,4-5,5; p <0,05).
ConclusionesSi bien los genotipos I/Ia y E de Chlamydia trachomatis fueron la causa más frecuente de infección respiratoria en neonatos mexicanos, el 80% de los genotipos F produjeron este padecimiento. En cambio, el genotipo D se asoció con el desarrollo de sepsis neonatal y el genotipo I/Ia con corioamnionitis.
One hundred thirty million Chlamydia trachomatis infections are reported worldwide each year. Nineteen serotypes of this pathogen can cause infection in pregnant women and neonates. The distribution of these genotypes in newborns with respiratory infections in Mexico is unknown.
Material and methodsWe tested 1062 bronchial lavage samples from neonates with respiratory distress syndrome for Chlamydia infection. The diagnosis of Chlamydia was made by plasmid detection with an in-house PCR assay, and genotypes were identified using a PCR-RFLP assay for the ompA gene.
ResultsThe genotyping of 40 strains identified 14 as I/Ia (35%), 13 as E (32.5%), 7 as D (17.5%), 5 as F (12.5%), and 1 as L2 (2.5%). The relative risk analysis showed that genotype D was associated with neonatal sepsis (RR, 5.83; 95% confidence interval [CI], 1.51-25.985; P<.02), while the I/Ia genotype was significantly associated with chorioamnionitis in the mother (2.8; 95% CI, 1.4–5.5; P<.05).
ConclusionsAlthough Chlamydia trachomatis genotypes I/Ia and E of were the strains involved most frequently in respiratory infections in Mexican neonates, 80% of patients with genotype F developed respiratory disease. In contrast, genotype D was associated with neonatal sepsis, and genotype I/Ia with chorioamnionitis.
Chlamydia trachomatis(C. trachomatis) es una bacteria intracelular obligada gramnegativa. Es la bacteria de transmisión sexual más prevalente en el mundo, reportándose cada año 131 millones de casos nuevos en adultos y adolescentes de 15 a 49 años1. Las gestantes pueden mantenerse asintomáticas durante meses, si bien pueden aparecer síntomas en cualquier momento del embarazo2. En el primer trimestre, la presencia de C. trachomatis previa a la interrupción del embarazo se ha asociado al desarrollo de endometriosis o salpingitis en el 20% de las gestantes afectadas2. En el segundo y tercer trimestre, su presencia se ha asociado al desarrollo de corioamnionitis, la rotura prematura de membranas (RPM), el parto pretérmino, el bajo peso al nacer e incluso la muerte fetal2,3. Tras el nacimiento, los neonatos pueden desarrollar neumonía (5-20%) y conjuntivitis (2-8%)4. Los genotipos que causan conjuntivitis con mayor frecuencia son el E y el G, seguidos del D, F, y K5,6. No obstante, la evidencia sobre los genotipos asociados al desarrollo de neumonía es escasa y de valor limitado7,8.
En México, la prevalencia de infección por C. trachomatis en embarazadas es del 6,73%9. Sin embargo, no se conocen los genotipos causantes de la infección, aunque se ha propuesto que el F puede ser el más frecuente en base a estudios realizados en mujeres infértiles10. En cuanto a los neonatos, la prevalencia de la infección en México es del 11,6%, con una tasa de transmisión vertical del 1,5% cuando se trata a la madre con azitromicina antes del parto11. A pesar de todo esto, no hay datos sobre la prevalencia de los genotipos que causan infección en neonatos, y solo un estudio realizado de Hernández Trejo et al.12 identificó el genotipo D en el tejido hepático de un recién nacido que falleció por causas desconocidas en el Instituto Nacional de Perinatología de la Ciudad de México. En este mismo estudio se detectó la presencia de ADN de C. trachomatis en más de un órgano en el 35,7% de los fallecimientos neonatales12.
El objetivo de nuestro estudio fue identificar los genotipos de C. trachomatis que causan infecciones respiratorias y conjuntivales en pacientes neonatales en una institución de salud terciaria en la Ciudad de México.
Materiales y métodosMuestras biológicasEstudio transversal descriptivo de 79 muestras de lavado bronquial obtenidas de neonatos atendidos entre el 1 de enero de 2016 y el 31 de diciembre de 2019 en una institución de salud de tercer nivel en la Ciudad de México. El estudio incluyó a neonatos con resultados negativos en las pruebas de detección de los patógenos respiratorios asociados con mayor frecuencia a la neumonía afebril, con eosinofilia (> 400 eosinófilos/mm3) y clínica sugestiva de infección por bacterias atípicas. Los criterios de exclusión fueron la falta de datos en el historial médico y el tratamiento antimicrobiano en el momento de la recogida de la muestra o en las 72 h precedentes. Las muestras recogidas se introdujeron en medio de transporte 2-SP para C. trachomatis para su conservación. Una vez llegaban al laboratorio, las muestras se procesaron para la extracción de ácidos nucleicos.
Definición de resultados, signos y síntomasEl diagnóstico de distrés respiratorio se basó en la presencia de aleteo nasal, quejido espiratorio, tiraje xifoideo, tiraje costal, disociación toracoabdominal, polipnea, agotamiento, y apnea; evidencia en la monitorización de hipoxia, hipercapnia, asfixia y acidosis, una presión de oxígeno (PaO2) inferior a 50mmHg; cianosis central en aire ambiente, o la necesidad de oxígeno suplementario para mantener un PaO2 superior a 50mmHg; características radiográficas consideradas típicas de la enfermedad (patrón reticulogranular difuso con broncograma aéreo u opacidades en vidrio esmerilado). El diagnóstico diferencial del distrés respiratorio incluyó la neumonía perinatal, el síndrome de distrés respiratorio, la taquipnea transitoria del recién nacido y el síndrome de aspiración de meconio. Se registraron las complicaciones posparto identificadas en las primeras 72 h de vida, incluyendo conjuntivitis y sepsis.
La infección respiratoria se consideró en neonatos con antecedentes de infección materna, rasgos sugestivos de corioamnionitis o RPM; que, tras el nacimiento, mostraron signos de fallo respiratorio, estertores, rasgos sugestivos de respuesta inflamatoria sistémica con aumentos en la frecuencia cardíaca y la respiratoria, cambios en la temperatura corporal o problemas hemodinámicos, con rasgos radiográficos de ocupación alveolar con apariencia parcheada y broncograma aéreo, rasgos sugestivos de respuesta inflamatoria sistémica, como leucocitosis, leucopenia o recuento plaquetario disminuido en el hemograma; resultados positivos de las pruebas de la proteína C reactiva y la procalcitonina o el hemocultivo, o identificación de alguna bacteria conocida como causante de neumonía atípica.
Aprobación para la investigación en seres humanosLas pruebas para el diagnóstico de la infección por clamidia se realizaron en los recién nacidos tras obtener el consentimiento informado firmado por los padres o tutores legales. El estudio fue aprobado por el Comité de Ética institucional (ID 212250–3120-10607–01-14) y se ajustó a los principios éticos de la institución, así como a los de la Declaración de Helsinki de 1975, según la enmienda del año 2000.
Extracción de ADN genómicoEl ADN genómico se obtuvo por el método de extracción con fenol-cloroformo. La integridad del material genético se verificó por electroforesis en gel de agarosa al 1%. La concentración y la pureza del ADN (μg/mL) se midieron con un espectrofotómetro (Nanodrop Biochrom™, GeneQuant™ 1300). El ADN obtenido se conservó a−20°C hasta su utilización.
Identificación de C. trachomatisSe utilizó un ensayo de reacción de polimerasa en cadena convencional para identificar la infección por C. trachomatis mediante la detección de su plásmido críptico. Para ello, se amplificó un fragmento de 241 pb de la región codificante 1 (CDS1) con los cebadores descritos por Mahony et al.13, KL1 5’-TCCGGAGCGAGTTACGAAGA-3’ y KL2 5’-AATCAATGCCC GGGATTGGT-3’. Mahony et al. ya han descrito previamente la mezcla de reacción y las condiciones de la PCR13. Como control negativo se utilizó ADN de células McCoy no infectadas y como control positivo ADN de C. trachomatis (ATCC: DUWC3X).
GenotipadoEl genotipado de C. trachomatis se llevó a cabo mediante análisis del polimorfismo de longitud de fragmentos de restricción amplificados por PCR (RFLP-PCR), según se ha descrito previamente10,14. Los perfiles de restricción se estudiaron mediante la electroforesis en gel de poliacrilamida al 12%, y el genotipo de las cepas de C. trachomatis se determinó comparando los resultados del análisis con una base de datos genética.
Secuenciación de ADNEn los casos en los que el ensayo RFLP no identificó ningún genotipo de C. trachomatis, se realizó un ensayo de PCR anidada. La identificación de estos genotipos requirió la amplificación del segmento variable 2 (VS-2) del gen ompA. Para ello, en primer lugar se amplificó un fragmento de 1000 pb correspondiente a la región entre los segmentos variables 1 y 2 con los cebadores publicados por Zheng et al.15 (CT1: 5′-TGA ACCAAGCCTTATGATCGACGGA-3′ y CT2: 5′-CGGAATTGTGCATTTACGTGA G-3′). A continuación, el producto de la amplificación sirvió de plantilla para la amplificación del segmento variable 2 utilizando los cebadores CT3: 5′-ACTTTGTTTTCGACCGTGTTTTG-3′ y CT4: 5′-GATTGAGC GTATTGGAAAGAAGC -3’. Estos cebadores amplificaron un fragmento de 416 pb. Las mezclas y condiciones de reacción fueron similares en ambos ensayos. La mezcla de la PCR contenía 1,75mM de MgCl2, 30 pM/μL de cada cebador, 160μM/μL de nucleótidos, 1 U/μL de polimerasa Taq (Promega), y 100 ng/μL de ADN. La reacción se llevó a cabo en un termociclador PTC-100 (MJ Research Inc) bajo las siguientes condiciones: desnaturalización a 94°C durante 5min, seguida de 30 ciclos de alineamiento consistentes en 1 minuto a 94°C, 1 minuto a 55°C y 1 minuto a 72° C y un ciclo final de extensión de 5 min a 72°C. Por último, el producto de 416 pb fue secuenciado con un analizador ABI PRISM 310 (PE Biosystems) empleando un kit de secuenciación de ADN BigDye (PE Biosystems) de acuerdo con las instrucciones del fabricante.
Análisis estadísticoSe obtuvo una muestra aleatoria de entre las muestras que resultaron positivas para C. trachomatis para realizar el genotipado. La selección se llevó a cabo generando números aleatorios entre el 1 y el número total de muestras positivas con una calculadora científica Casio FX-82MS (Casio Computer Co, Ltd; Tokio, Japón).
El análisis estadístico se realizó con el software SPSS Statistics, versión 24.0 parar Windows (IBM Corp., Armonk, NY, EE. UU.), con la prueba exacta de Fisher. La magnitud de la asociación entre variables se ha expresado en términos de riesgos relativos (RR) con sus correspondientes intervalos de confianza (IC) del 95%, empleándose la prueba t de Student para los datos paramétricos y las pruebas de Kruskal Wallis y U de Mann-Whitney para los datos no paramétricos. Los valores de p inferiores a 0,05 se consideraron significativos.
El tamaño muestral requerido para detectar una asociación entre las variables clínicas y la infección neonatal por C. trachomatis se calculó con la siguiente fórmula:
n=N Z2pq /d2 (N−1)+Z2pq
El número de muestras positivas para C. trachomatis fue 363, con un margen de error del 15% debido a la existencia de cepas de C. trachomatis carentes del plásmido; con lo que según la fórmula aplicada, una selección de 39 muestras sería suficiente para explorar las asociaciones.
ResultadosPrevalencia de la infección por C. trachomatis en neonatosEntre el 1 de enero de 2016 y el 31 de diciembre de 2019, se analizó un total de 1062 muestras de neonatos con síndrome de distrés respiratorio para la detección de C. trachomatis. Hubo 363 muestras positivas para ADN de C. trachomatis, lo que corresponde a una prevalencia del 34,18%.
De estos 363 neonatos, 207 eran varones y 156 mujeres. El tipo de parto más frecuente fue por cesárea (65,3%) y el 34,7% fueron vaginales (p <0,05). En el 16,8% (61/363), había antecedente de RPM y en el 14,9% (54/363) de corioamnionitis en madres infectadas por Chlamydia (datos no publicados).
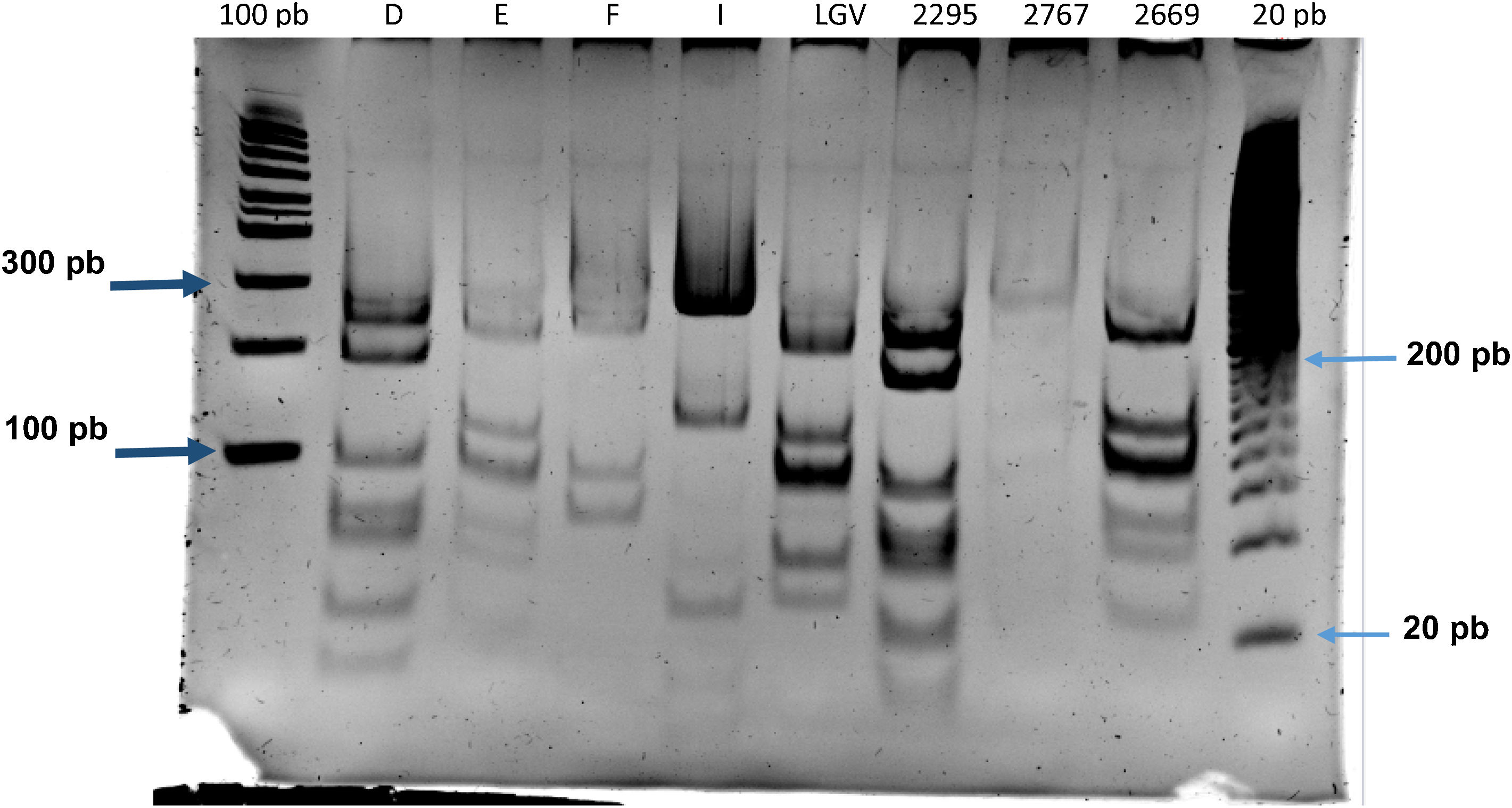
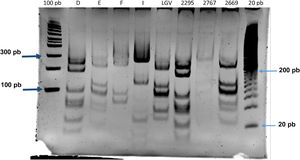
Identificación de genotipos de C. trachomatisDel total de 363 muestras positivas para C. trachomatis, 79 (21,8%) se seleccionaron aleatoriamente para el genotipado de la cepa implicada en la infección mediante PCR-RFLP. La figura 1 muestra los fragmentos obtenidos en el ensayo de PCR-RFLP de cepas de clamidia y las muestras positivas para clamidia. En 3 muestras no se pudo identificar el genotipo, por lo que se obtuvo y analizó la secuencia de nucleótidos del segmento variable VS-2 del gen ompA. Las secuencias se compararon con los genotipos registrados en la base de datos GenBank de la National Center for Biotechnology Information (NCBI), identificándose 2 de ellas como genotipo E y una como genotipo L2 (fig. 2). El genotipado de las 79 muestras solo consiguió identificar el genotipo de 40 cepas (tabla 1): 14 como I/Ia (35%), 13 como E (32,5%), siete como D (17,5%), cinco como F (12,5%), y una como L2 (2,5%).
Polimorfismos de longitud de fragmentos de restricción del gen ompA en cepas de C. trachomatis aisladas de muestras de aspirado bronquial de recién nacidos con distrés respiratorio. Tipo de cepa (D, E, F, I); muestra de aspirado bronquial (2295, 2767, 2669), marcador de peso molecular (100 pb y 20 pb).
Asociación entre el genotipo de C. trachomatis y las características clínicas del neonato
| Clínica | D | E | F | I | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Sí | No | RR% (IC 95%) | p | Sí | No | RR% (IC 95%) | p | Sí | No | RR% (IC 95%) | p | Sí | No | RR% (IC 95%) | p | |
| Sexo | ||||||||||||||||
| Varón | 4 | 21 | 0,8 (0,21-3,1) | NS | 9 | 16 | 1,35 (0,5-3,6) | NS | 4 | 21 | 2,4 (0,3-19,5) | NS | 8 | 17 | 0,8 (0,3-1,9) | NS |
| Mujer | 3 | 12 | 4 | 11 | 1 | 14 | 6 | 9 | ||||||||
| Tipo de parto | ||||||||||||||||
| Vaginal | 2 | 6 | 1,6 (0,4-6,8) | NS | 3 | 5 | 1,2 (0,4-3,4) | NS | 2 | 6 | 2,7 (0,5-13,4) | NS | 1 | 7 | 0,31 (0,05-2,0) | NS |
| Cesárea | 5 | 27 | 10 | 22 | 3 | 29 | 13 | 19 | ||||||||
| Sepsis | ||||||||||||||||
| Sí | 5 | 7 | 5,8 (1,5-26) | 0,017 | 3 | 9 | 0,7 (0,23-2,1) | NS | 1 | 10 | 1,6 (0,3-8,2) | NS | 2 | 10 | 0,39 (0,1-1,5) | NS |
| No | 2 | 26 | 10 | 18 | 4 | 25 | 12 | 16 | ||||||||
| BPD | ||||||||||||||||
| Sí | 5 | 15 | 2,5 (0,6-11,4) | NS | 10 | 10 | 3,3 (1,1-10,3) | 0,04 | 1 | 19 | 0,25 (0,06-3,8) | NS | 4 | 16 | 0,4 (0,15-1,1) | NS |
| No | 2 | 18 | 3 | 17 | 4 | 16 | 10 | 10 | ||||||||
| Neumonía | ||||||||||||||||
| Sí | 5 | 21 | 1,35 (0,3-6,07) | NS | 6 | 20 | 0,46 (0,19-1,1) | NS | 4 | 19 | 3,0 (0,36-24,1) | NS | 10 | 16 | 1,4 (0,52-3,5) | NS |
| No | 2 | 12 | 7 | 7 | 1 | 16 | 4 | 10 | ||||||||
| Conjuntivitis | ||||||||||||||||
| Sí | 2 | 7 | 1,4 (0,32-6,0) | NS | 3 | 6 | 1,0 (0,36-3,0) | NS | 1 | 8 | 0,86 (0,1-6,8) | NS | 3 | 6 | 0,9 (0,3-2,7) | NS |
| No | 5 | 26 | 10 | 21 | 4 | 27 | 11 | 20 | ||||||||
| Corioamnionitis | ||||||||||||||||
| Sí | 1 | 4 | 1,7 (0,18-7,8) | NS | 0 | 5 | 1,6 (1,2-2,0) | NS | 0 | 5 | 1,7 (1,0-1,34) | NS | 4 | 1 | 2,8 (1,4-5,5) | 0,043 |
| No | 6 | 29 | 13 | 22 | 5 | 30 | 10 | 25 | ||||||||
| RPM | ||||||||||||||||
| Sí | 4 | 12 | 2,0 (0,52-7,8) | NS | 3 | 13 | 0,45 (0,15-1,4) | NS | 1 | 15 | 0,38 (0,05-3,1) | NS | 8 | 8 | 2,0 (0,86-4,7) | NS |
| No | 3 | 21 | 10 | 14 | 4 | 20 | 6 | 18 | ||||||||
DBP: displasia broncopulmonar; IC: intervalo de confianza; NS: no significativo; RPM: rotura prematura de membranas; RR: riesgo relativo.
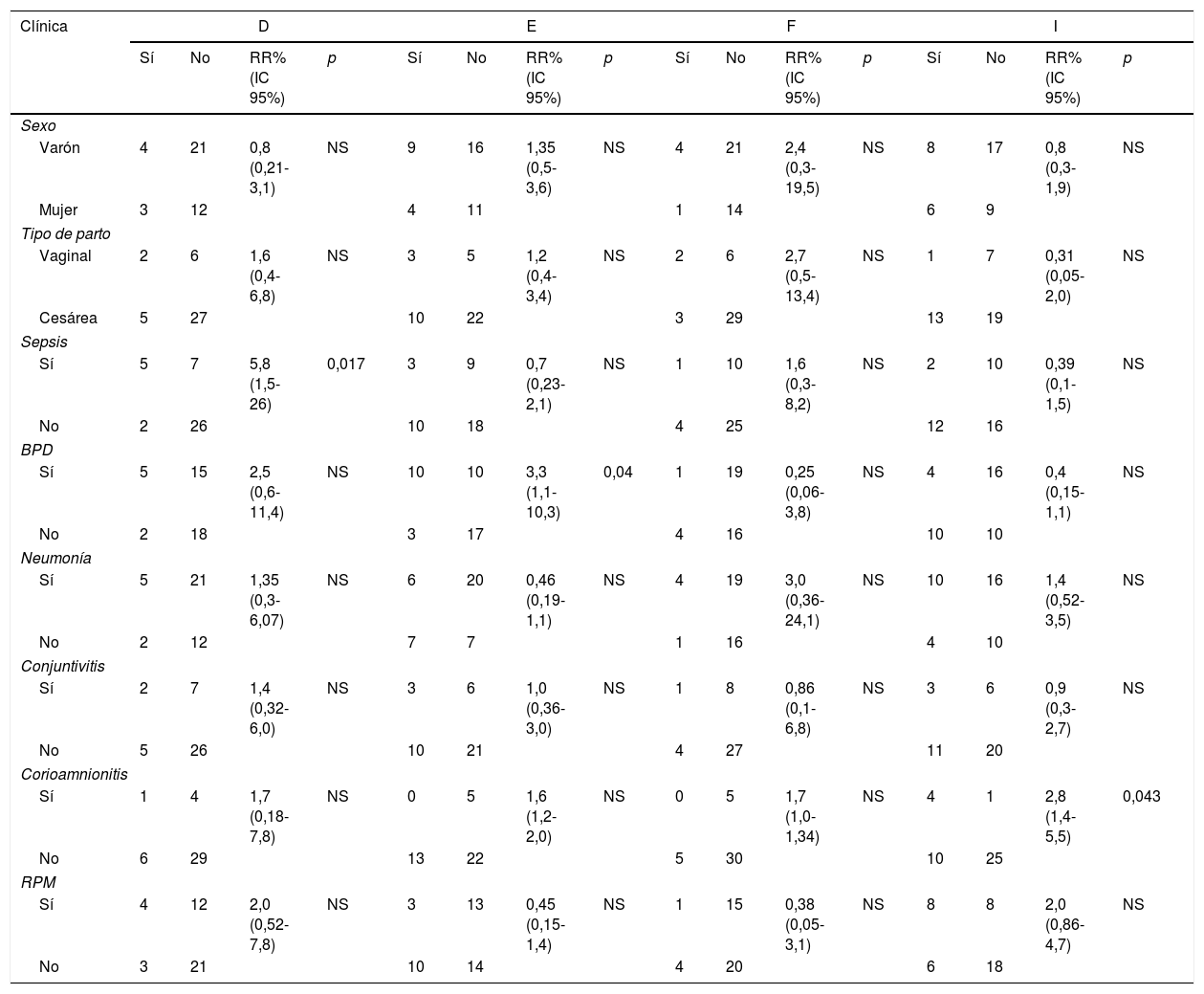
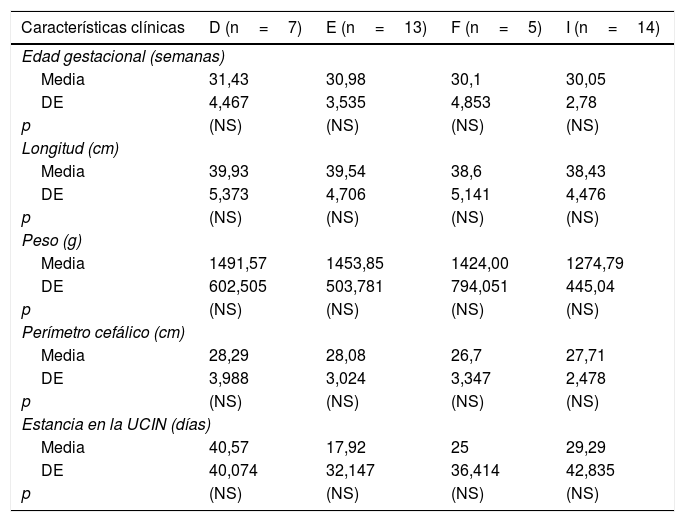
De los 40 del total de 363 neonatos (11%) en los que se identificó el genotipo de C. trachomatis implicado, 25 eran varones y 15 mujeres, y 8 habían nacido por parto vaginal y 32 por cesárea (tabla 1). Al nacimiento, la edad gestacional media fue de 30,6± 3,87 semanas, y la longitud, peso y perímetro cefálico medios fueron 39±4,9cm, 1411±586,4g y 27,7±3,21cm, respectivamente (tabla 2). No se encontró una asociación significativa entre ninguna de estas variables y el genotipo de C. trachomatis causante de la infección. De manera similar, algunas manifestaciones clínicas, como la conjuntivitis o la neumonía, no se asociaron a algunos de los genotipos identificados. No obstante, se observó un riesgo relativo de sepsis cuatro veces mayor en neonatos infectados por el genotipo D (RR=5,83; IC 95%: 1,51-25,985; p <0,02). Además, la displasia broncopulmonar fue más frecuente en neonatos infectados por el genotipo E (RR=3,3; IC 95%: 1,1-10,3; p <0,05), y el genotipo I/Ia se asoció significativamente con el desarrollo de corioamnionitis en la madre (RR=2,8; CI 95%: 1,4-5,5; p <0,05).
Diferencias en las medidas antropométricas y las variables clínicas en neonatos con distrés respiratorio según el genotipo de C. trachomatis
| Características clínicas | D (n=7) | E (n=13) | F (n=5) | I (n=14) |
|---|---|---|---|---|
| Edad gestacional (semanas) | ||||
| Media | 31,43 | 30,98 | 30,1 | 30,05 |
| DE | 4,467 | 3,535 | 4,853 | 2,78 |
| p | (NS) | (NS) | (NS) | (NS) |
| Longitud (cm) | ||||
| Media | 39,93 | 39,54 | 38,6 | 38,43 |
| DE | 5,373 | 4,706 | 5,141 | 4,476 |
| p | (NS) | (NS) | (NS) | (NS) |
| Peso (g) | ||||
| Media | 1491,57 | 1453,85 | 1424,00 | 1274,79 |
| DE | 602,505 | 503,781 | 794,051 | 445,04 |
| p | (NS) | (NS) | (NS) | (NS) |
| Perímetro cefálico (cm) | ||||
| Media | 28,29 | 28,08 | 26,7 | 27,71 |
| DE | 3,988 | 3,024 | 3,347 | 2,478 |
| p | (NS) | (NS) | (NS) | (NS) |
| Estancia en la UCIN (días) | ||||
| Media | 40,57 | 17,92 | 25 | 29,29 |
| DE | 40,074 | 32,147 | 36,414 | 42,835 |
| p | (NS) | (NS) | (NS) | (NS) |
DE: desviación estándar; NS: no significativo; UCIN: unidad de cuidados intensivos neonatales.
C. trachomatis se ha asociado a varias patologías que afectan al embarazo, el desarrollo fetal y el recién nacido2,16,17, y puede contribuir al nacimiento pretérmino, la corioamnionitis, la RPM e incluso la muerte neonatal, aunque esto último continúa siendo objeto de debate. En nuestro estudio, la edad gestacional media de los 363 neonatos con resultados positivos para infección por clamidia fue de 31,5±4,1 semanas, y hubo antecedente de corioamnionitis en el 15% y de RPMP en el 17%.
El nacimiento pretérmino y el bajo peso al nacer se asocian con frecuencia a la infección por C. trachomatis3,16,17, como demuestra nuestro estudio, en el que la mayoría de los neonatos nacieron antes de las 32 semanas de gestación y con peso bajo. La prematuridad está reconocida como la causa más importante de morbimortalidad perinatal a nivel mundial, y es responsable del 27% de los casi cuatro millones de muertes neonatales reportadas anualmente. La enfermedad pulmonar crónica y las infecciones son dos de los factores de riesgo de mortalidad perinatal más importantes. La tasa de mortalidad neonatal media reportada por el Instituto Nacional de Perinatología en estos cuatro años de investigación fue de 19,9 defunciones por cada 1000 nacidos vivos. En nuestro estudio, hubo 13 defunciones entre los 363 recién nacidos con infección por clamidia (3,6%) (el genotipo solo se identificó en un caso, y fue el E). Esto sugiere que C. trachomatis puede ser una causa importante de mortalidad perinatal, como ya han propuesto otros autores18.
La prevalencia global de la infección por C. trachomatis en mujeres embarazadas varía entre el 6,7% y el 18%16–21. En México, es del 4,7% en adolescentes embarazadas y del 2% en gestantes adultas9. La frecuencia reportada de la infección neonatal varía entre el 2% y el 40% a nivel mundial6,22,23 y entre el 11,6% y el 45% en México11,24. En nuestro estudio, la prevalencia observada de infección por C. trachomatis fue del 35%.
Se ha descrito que entre el 30% y el 50% de los recién nacidos con infección por clamidia desarrollan conjuntivitis y entre el 10% y el 20% neumonía2–4. No obstante, en nuestro estudio la conjuntivitis no fue frecuente, ya que solo se desarrolló en 34 de los 363 neonatos infectados (9,4%). El genotipo solo se pudo identificar en 9 de estos casos (2,5%), y los más frecuentes fueron el E (0,8%) y el I/Ia (0,8%) (3 casos cada uno).
La frecuencia de neumonía fue ligeramente mayor, del 24,8% (90/363). El genotipo I/Ia fue el identificado con mayor frecuencia en estos casos (2,75%), seguido de los genotipos E (1,4%), F (1,1%) y D (1,1%). Cabe mencionar que 4 de los 5 pacientes en los que se identificó el genotipo F desarrollaron neumonía.
La estancia hospitalaria media de los recién nacidos fue de 55 días, y fueron diagnosticados de infección del tracto respiratorio causada por microorganismo atípico. La estancia media prolongada podría deberse a las complicaciones intrínsecas de la prematuridad y de la enfermedad. La infección cursó con displasia broncopulmonar (DBP) en el 65% de los pacientes. Carrera-Muiños et al.23 reportaron resultados similares en cuanto a la estancia hospitalaria en neonatos con infección por clamidia.
En México no se ha investigado previamente qué genotipos de C. trachomatis causan infección en neonatos. Estudios previos han demostrado que el genotipo F es más frecuente en mujeres infértiles y hombres cuyas parejas son estériles en la Ciudad de México10,25. En Guadalajara, México, los genotipos E, F, y D son los más frecuentes en mujeres infértiles, mientras que los genotipos F y E son más comunes en gestantes21. En nuestro estudio, de las 40 muestras en las que se identificó el genotipo de clamidia causante de infección en recién nacidos, los genotipos más frecuentes fueron el I/Ia (35%) y el E (32,5%), seguidos del D (17,5%), el F (12,5%), y el L2 (2,5%).
Aunque en el presente estudio no se pudo demostrar que ningún genotipo de C. trachomatis fuera la causa única de alguna condición en particular, sí se pudieron discernir algunas tendencias. El genotipo D ya se había descrito en México como causante de sepsis neonatal. Este genotipo fue detectado en una muestra de tejido hepático de un neonato fallecido con clínica de sepsis neonatal sin aislamiento bacteriano y con antecedentes de corioamnionitis y RPM de 8 h de duración12. En nuestro estudio, el genotipo D fue 4,6 veces más frecuente en neonatos que desarrollaron sepsis (p <0,04). La sepsis por C. trachomatis no es nada nuevo; en 2016, un estudio realizado en Ghana encontró una prevalencia de sepsis del 33,7% y RPM (p <0,001) en recién nacidos de madres infectadas por C. trachomatis26. No obstante, los autores no identificaron los genotipos causantes de la enfermedad.
A nivel internacional, en Argentina se ha encontrado una proporción del genotipo D entre el 5,3% y el 7,9% en casos de conjuntivitis neonatal5, mientras que en Chile y China, este genotipo estaba implicado con mayor frecuencia en casos de neumonía neonatal, con proporciones del 11,8% y el 12,1%, respectivamente7,8. En el presente estudio, estas enfermedades estaban presentes en el 22,2% y el 19,2% de los casos, respectivamente.
El genotipo D es uno de los más invasivos porque tiene una citotoxina que puede lisar las células de la mucosa genitourinaria, como demostró el estudio realizado por Stephens et al.27 Esta citotoxina está codificada por las secuencias genéticas CT165 a CT168, dando lugar a una proteína cuya secuencia de aminoácidos presenta una homología significativa con las de las citotoxinas de Clostridium difficile28. Por añadidura, causa alteraciones en el citoesqueleto de las células huésped por medio de su actividad como glucosiltransferasa, modificando moléculas reguladoras intracelulares, como las proteínas de unión al GTP de la familia Rho/Rac29–31. Estos cambios citoesqueléticos pueden destruir la membrana celular y de este modo promover el desarrollo de sepsis.
Otro hallazgo importante en nuestro estudio fue la identificación de una secuencia de nucleótidos correspondiente a una cepa causante de linfogranuloma venéreo (genotipo L2 de C. trachomatis) en una muestra neonatal. La madre de este recién nacido no desarrolló signos ni síntomas de linfogranuloma venéreo. La prevalencia del genotipo L2 en neonatos no se ha establecido; solamente se ha descrito en una ocasión por Beem et al.32, que detectaron anticuerpos contra el serotipo L1 de C. trachomatis en 18 recién nacidos entre 20 con neumonía. Asimismo, en varios estudios se detectó la infección por el genotipo L2, genotipo en hombres heterosexuales y homosexuales y en mujeres que no desarrollaron linfogranuloma venéreo10,33, lo que sugiere que esta es una variante del genotipo L2, posiblemente la L2f. Las secuencias de nucleótidos de la variante del genotipo L2 obtenidas en nuestro estudio mostraron deleciones, sustituciones e inserciones en las regiones VS1 y VS2 del gen ompA.
Por otro lado, el genotipo E es el más prevalente en neonatos con neumonía en Chile (47,1%)7 y China (42,4%)8 y en neonatos con conjuntivitis en Argentina (72,4%)5 y Hungría (48%)6. En nuestro estudio, el genotipo E fue el segundo en frecuencia en recién nacidos con neumonía (23%) y se identificó en un único caso de conjuntivitis neonatal (11,1%). En cambio, se observó una tendencia en la que el 50% de los recién nacidos infectados por este genotipo desarrollaron DBP. No obstante, la etiología de DBP es multifactorial y puede implicar factores como la ventilación mecánica prolongada, concentraciones altas de oxígeno inspirado, corioamnionitis o sepsis o el grado de prematuridad, entre otros. Se necesita investigar más para determinar si el genotipo E se asocia a un grado mayor de inmadurez pulmonar, ya que en el presente estudio no se encontró asociación con el desarrollo de sepsis o la corioamnionitis. En Argentina, el genotipo E estaba implicado en el 7,9% de los casos de conjuntivitis neonatal5. En nuestro estudio, el 33,3% de los recién nacidos infectados por este genotipo desarrollaron la enfermedad.
En cuanto al genotipo F, encontramos una frecuencia menor de infección en neonatos comparada con la frecuencia observada en mujeres infértiles y hombres atendidos en el Instituto Nacional de Perinatología en la Ciudad de México10,25. Estudios en Chile y China han reportado proporciones de casos de neumonía por C. trachomatis correspondientes a este genotipo de 17,6% y 42,4%, respectivamente7,8. En nuestro estudio encontramos una proporción de 17,4%, similar a la de Chile. No obstante, cabe destacar que de los 5 neonatos infectados por el genotipo F, 4 desarrollaron neumonía (80%), lo que hace a este genotipo particularmente interesante como posible causa principal de neumonía. En el presente estudio no se pudo demonstrar una asociación con la neumonía, quizá por el escaso número de muestras que fueron positivas para este genotipo. La conjuntivitis causada por el genotipo F no se ha descrito con frecuencia en neonatos5,6. En este sentido, nuestros hallazgos fueron consistentes con la literatura previa.
En lo tocante al genotipo I/Ia, se ha reportado una prevalencia de 1,3% entre los neonatos con conjuntivitis en Argentina5. En nuestro estudio, se detectó el genotipo I/Ia en el 33,3% de los recién nacidos con conjuntivitis. La frecuencia del genotipo I/Ia como causa de neumonía no se ha reportado hasta la actualidad. En nuestro estudio, detectamos este genotipo en el 38,5% de los neonatos con neumonía.
Otro hallazgo interesante de nuestro estudio es que el genotipo I/Ia se asoció a un aumento de 2,8 veces estadísticamente significativo en el riesgo relativo de corioamnionitis; no hay datos previos sobre la prevalencia de los genotipos causantes de corioamnionitis. No obstante, en Estados Unidos, Geisler et al.34 han descrito una asociación entre el genotipo I/Ia y la raza negra en las mujeres, con una prevalencia del 16%, mientras que en mujeres infértiles en México se ha descrito una prevalencia del 4,2% al 6%10. Más allá de estos países, se ha descrito una prevalencia del 4,8% en Senegal, 2,1% en Costa Rica, y 16% en Guadalupe, en la región del Caribe35–37.
ConclusiónAunque los genotipos I/Ia y E de C. trachomatis fueron los implicados más frecuentemente en las infecciones del tracto respiratorio en neonatos mexicanos, el 80% de los pacientes con el genotipo F desarrollaron patología respiratoria. En cambio, el genotipo D se asoció al desarrollo de sepsis neonatal sepsis, y el genotipo I/Ia al desarrollo de corioamnionitis.
FinanciaciónLos autores no recibieron financiación para el desarrollo de este proyecto.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.