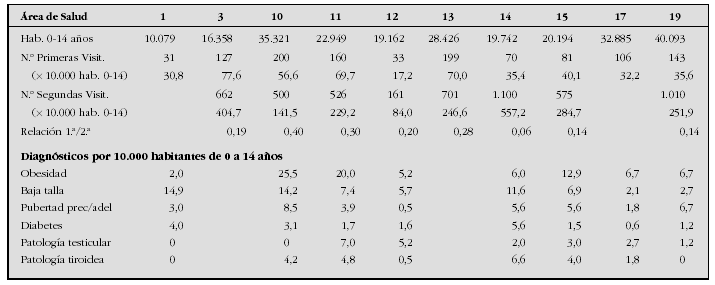
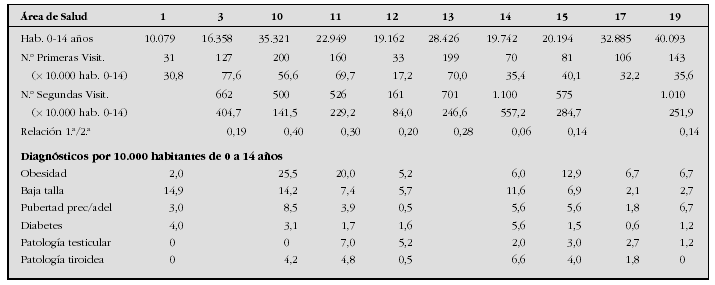
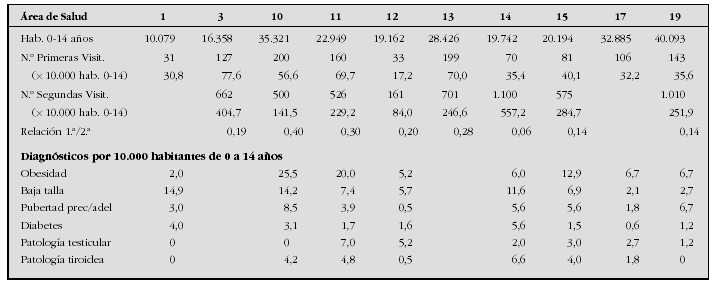
Introducción
Todo proceso capaz de limitar, interrumpir o interferir durante la vida intrauterina el potencial de crecimiento del feto provocará un crecimiento intrauterino retardado (RCIU)1,2.
El RCIU es un problema muy importante de salud pública en la mayoría de los países desarrollados. Las causas son múltiples e interactivas y su importancia relativa varía con el tiempo, lugar y población3,4.
El crecimiento fetal debe estar controlado a nivel celular y tisular por los nutrientes proporcionados y/o por factores activos localmente por ejemplo los IgF5,6, la GH placentaria7,8 y otros factores de crecimiento. El hecho de que factores de crecimiento de origen placentario: EGFR (receptor del factor de crecimiento epidérmico), PDGF (factor de crecimiento derivado de las plaquetas), IGF (factor de crecimiento similar a la insulina), TGFα (factor de crecimiento transformador tipo α) actúen localmente favoreciendo el desarrollo y diferenciación de la placenta, y que desde el punto de vista general, actúen se centra en competencias inmunológicas, hacen que se otorgue a la placenta funciones importantes en el desarrollo y regulación del crecimiento fetal.
Todos los tejidos fetales incluida la placenta expresan mRNA mensajero de IGFI9,10 La concentración de IGFI en plasma materno correlaciona con el crecimiento fetal11,12 IGFI, IGFII e insulina materna aunque no cruzan la placenta, por lo que no tienen efecto directo en el crecimiento fetal, si que poseen influencia directa en la función placentaria13,14.
Existen en la bibliografía diferentes trabajos confirmando que la concentración de IGFI en sangre fetal y cordón umbilical se correlaciona con el tamaño del feto. Por otra parte, cuando se trasplantan embriones de ratón que producen gran cantidad de IGFI en embriones de ratón normal se produce un estímulo del crecimiento superior a cuando al ratón normal se le trasplantan embriones con una secreción escasa en IGFI15.
El factor de crecimiento epidérmico (EGF), es un polipéptido de cadena única, con un peso molecular de aproximadamente 6.000, estimula la proliferación y diferenciación en muchas clases de células y tejidos16-18. El EGFR, glicoproteina de 170.000 dalton, ha sido también intensamente estudiada y localizado en el cromosoma 17, en la región p13-q22.
El receptor de crecimiento epidérmico EGFR es un receptor celular de superficie, con un dominio externo, un dominio transmembrana y un dominio intracelular con actividad tirosinkinasa. Pertenece al grupo de receptores transmembrana homólogos con actividad tirosinkinasa como c-erbB2, c-erbB3 y c-erb B4. Los ligandos de este receptor incluyen el EGF, factor alfa de crecimiento transformación (TGFα, la amphiregulina, cripto EGF y EGF que se une a la heparina. De éstos, EGF y TGFα están implicados en la carcinogénesis. In vitro, EGFR y sus ligandos están implicados en la transformación maligna vía autocrina y paracrina del mecanismo del crecimiento. In vivo, la sobrexpresión de EGFR se ha reportado en numerosos canceres, especialmente en el carcinoma de células escamosas, y se cree que se relacione con el mal pronóstico. Sin embargo, existen publicaciones que muestran que algunos tejidos normales expresan EGFR a más altas concentraciones que sus homólogos malignos19,20.
El mecanismo primario que regula el nivel de EGFR es la abundancia de su mRNA. Está codificado por un gen con dos transcriptos de RNA de 10,5 kb y 5,8 kb Parece que cada tránscripto se expresa de forma diferente durante diversos estadios del desarrollo placentario, y además hay otro tránscripto putativo del EGFR de 1,8 kb que codifica solo el dominio de unión del receptor21,22.
Se han realizado numerosos estudios con EGF y EGFR, y, los aspectos biológicos han alcanzado mucho interés. El importante papel de EGF se ha demostrado en el crecimiento fisiológico y neoplásico de tejido mamario y en la espermatogénesis del ratón. Se ha demostrado que el nivel de EGF se incrementa en glándula submandibular y plasma durante el embarazo y que su deficiencia causa aborto y RCIU en ratón. También se ha demostrado que EGFR y su mRNA se incrementa durante el embarazo en placenta humana y en células trofoblásticas aisladas y sugieren que EGF juegue un papel en el desarrollo fetoplacentario23.
Se expresa fuertemente en placenta humana y se incrementa la expresión a lo largo de la gestación. La diferente expresión de EGFR y sus ligandos sugieren un papel diferente de los ligandos de EGF a lo largo de la gestación. Al comienzo parece estimular la proliferación y poder de invasión del citotrofoblasto implicado en la implantación. Mas tarde, a partir de la 6.ª semana EGF estimula la diferenciación del citotrofoblasto a sincitiotrofoblasto.
Objetivo
El objetivo de este trabajo ha sido el análisis de la expresión de los genes del factor de crecimiento similar a la insulina tipo 1 (IGFI) y el receptor de crecimiento epidérmico (EGFR) en tejido placentario de gestaciones normales y de retrasos de crecimiento intrauterino (RCIU) en un intento de aportar explicaciones a la etiología del proceso. Se ha utilizado la RT-PCR relativa como método más sensible del estudio comparativo.
Material
Grupo control: Constituido por 42 muestras de placenta procedentes de gestaciones normales, sin antecedentes familiares ni personales de interés.
Grupo CIR: Se recogieron 31 muestras de tejido placentario de gestaciones con fetos CIR en los que se descartó el origen genético, malformativo, etc. del retraso de crecimiento.
Método
Recogida de la muestra
En todas las ocasiones, la muestra placentaria se obtuvo inmediatamente después de producirse el período expulsivo, en el propio paritorio o en quirófano cuando la finalización del parto fue por cesárea.
Se tomaron dos muestras de unos 3 mm3 (aproximadamente 30-35 mg) Con ayuda de tijeras de disección, se profundizó 1 o 2 cm de la superficie de la placenta para evitar la capa más superficial donde, por el desgarro generado al desprenderse del endometrio, las células pueden estar dislaceradas y, por consiguiente, el RNA mas degradado. El lugar de la muestra se eligió a unos 2 cm desde la inserción del cordón umbilical e intentado evitar las zonas de gran acúmulo sanguíneo.
Rápidamente se lavaron en Na2Cl estéril y frío para hemolizar hematíes y después se sumergieron en un protector del RNA (RNA later. Ambion) que permite conservarlo hasta 1 semana a temperatura ambiente. No obstante, siempre que fue posible, se procedió seguidamente a la extracción del RNA, o se congeló hasta su extracción en la misma jornada laboral.
Extracción del RNA: Se siguieron las instrucciones del fabricante del kit Qiagen. Este método está basado en hacer pasar a través de una matriz incluida en unas columnas, las células del tejido que previamente ha sido homogeneizado. El RNA queda atrapado en dicha matriz y, después de una serie de lavados, se libera y eluye en agua destilada o TE. Se comprueba la cantidad y calidad del RNA extraído en un gel de agarosa al 2 % y se procede seguidamente a la reacción de RT-PCR y el resto de la muestra se conserva a 20 °C.
RT-PCR. Se ha utilizado el kit Ambion siguiéndose las indicaciones del fabricante.
Se mezclan aproximadamente 1-2 mg de RNA con 4 ml de DNTP y 2 ml de oligodecámeros en un pequeño volumen de agua destilada estéril y se colocan a 70-85 °C durante 3 min. e inmediatamente se para la reacción introduciendo el tubo en hielo. Se añade un inhibidor del la RNAsa y el enzima la transcriptasa reversa con su buffer. Permanecerá durante 1 hora a 42-44 °C y luego a 92 °C durante 10 min, para detener la acción del enzima. En este punto, en el tubo ya tenemos cDNA que podremos conservar casi indefinidamente a 20 °C o continuar con la reacción de la PCR.
RT-PCR relativa
La RT-PCR relativa compara la abundancia de transcripto entre múltiples muestras por coamplificación de un control interno con el cADN en estudio, lo que permite una normalización para diferentes cantidades de ARN de las muestras. Para ello se utiliza QuantumRNA Competimer (Ambion). Se utiliza con el control interno 18srRNA como ideal para la mayoría de los experimentos, así se diseña una PCR múltiplex con los primers de 18s, Competimer y específicos del gen en estudio25,26.
Primers IGFI: 5'- CACGGCTGGACCGGAGACGC-3' 5'- GACCCAGAAGGAAGTACATTTG-3'
EGFR: 5'- GGAGGAAAAGAAAGTTTGCCAAGG-3' 5'- GAAATTTACAGGAAATCCTGCATGG-3'
La PCR se realizó en un volumen final de 25 ml con 1-2 ml de cDNA, 1,2 U de Taq polimerasa, 1-2 ml de la mezcla 18S/competimer y 1-2,5 ml del primer específico.
El programa consistió en 5 min. a 95 °C, 30 ciclos de 20 s a 94 °C, la hibridación fue a 59 °C durante 30 seg. Y la extensión de 72 °C durante 40 seg. seguidos de una extensión final de 5 min a 72 °C.
El producto PCR se hizo correr en un gel de agarosa al 2 % y los resultados se analizaron con un programa informático de análisis de geles que compara áreas de las bandas.
Análisis estadístico
Se utilizó el análisis comparativo de proporciones entre la intensidad de la banda del control interno y la muestra aplicando el test x2 de Pearson. Se consideró significativa una p < 0,05.
Resultados
La RT-PCR relativa26 es un método mas sensible que Northern blot o protección de nucleasas para la valoración de la expresión génica. Es capaz de detectar transcriptos moderadamente expresados, incluso con muy pocas células. La RT-PCR requiere que los primers para el gen en estudio sean compatibles con los del control interno y que la tasa de amplificación de ambos genes esté dentro de un rango lineal. Estas condiciones pueden resultar difíciles de optimizar dado que los controles internos comunes se expresan típicamente a un nivel mayor que el tránscripto en estudio.
Teóricamente, la cuantificación relativa por RT-PCR puede establecer diferencias en la abundancia de un tránscripto particular entre distintas muestras si se controla la cantidad de cDNA que se utilice, y si se usa el número de ciclos en la PCR necesarios sin llegar a sobrepasar la fase exponencial de la amplificación.
Como control interno se ha elegido el RNA ribosómico 18S (rRNA). Sin embargo, como se trata de un tránscrito extraordinariamente abundante, es necesario frenar su expresión con un competidor que amplifique con una pareja de primers similares especialmente modificados. Ajustando el porcentaje de 18S/competidor se logra que la coamplificación de lugar a una intensidad de bandas del control y del gen en estudio idéntica.
En gel de electroforesis de la figura 1, en la línea 1 corrió el marcador de peso molecular φ X174 RF DNA Hinf. III. En la línea 2 se optimizó la proporción 18S/competidor con la expresión de IGFI normal. En el carril 3 se compara control interno con EGFR.
Figura 1. 18s: 495 pb. IGFI: 289 pb. EGFR: 363 pb.
En la figura 2 se muestran los resultados de la amplificación de IGFI de tres placentas CIR (carriles 2, 3 y 4) evidentemente disminuidas en comparación con la de una placenta normal (carril 5). En la línea 1 el marcador de peso molecular φ X174 RF DNA Inf. III.
Figura 2. Línea 1: marcador f X174 RF DNA Hinf. III. Control interno 18S a 495 pb e IgFI a 289 pb.
En la figura 3, de igual modo, en las líneas 2, 3 y 4 son muestras de placentas CIR y la 5 es una placenta de gestación normal. No existen diferencias en la expresión entre ellas.
Figura 3. Línea 1: marcador f X174 RF DNA Hinf. III. Control interno 18S a 495 pb e EGFR a 363 pb.
El programa informático de análisis de geles, además de otros parámetros determina la altura exacta de la banda en comparación con el marcador de peso molecular, la intensidad de la tinción por bromuro de etidio de la banda, el área de la misma, y posteriormente, halla la razón entre el valor de la banda de 18S y el valor de la banda de IGFI o EGFR de cada muestra.
Expresión de IGFI
Se determinó en cada muestra la razón entre el área de la banda del control interno e IGFI y se consideró como valor de corte 1. Es decir que aquellas muestras con valores de 1 o más de 1 se tomaron como pertenecientes a una expresión disminuida de IGFI. Aquellas placentas cuyo cociente era inferior a 1 fueron consideradas con expresión de IGFI normal (figs. 4 y 5).
Figura 4. Agrupación de las muestras según el cociente expresión de 18S/expresión IGFI. Línea gris claro: placentas normales. Línea gris oscuro: placentas RCIU.
Figura 5. Expresión IGFI.
Como puede apreciarse en la figura 5, y considerando las columnas gris claro los recién nacidos normales y las gris oscuro los RCIU, se calculó que con una p significativa de 0,008 las placentas procedentes de gestaciones RCIU presentan una disminución de expresión de IgFI.
Expresión de EGFR
En la figuras 6 y 7 del mismo modo se muestran los resultados obtenidos al analizar la expresión de EGFR frente al control 18S. No se han encontrado diferencias significativas entre la expresión de este gen entre unas y otras.
Figura 6. Agrupación de las muestras según el cociente expresión de 18S/expresión EGFR. Línea gris claro: placentas normales. Línea gris oscuro: placentas RCIU.
Figura 7. Expresión de EGFR.
Conclusiones
Hemos querido estudiar dentro de las posibles causas del retraso de crecimiento fetal la expresión de factores de crecimiento, para lo cual se eligieron IGFI por ser un factor que se produce en la propia placenta, que como el resto de IGF no la atraviesan y con evidencias claras de que son responsables de la regulación de crecimiento fetal y placentario por lo que se asume su origen en la unidad fetoplacentaria y no a la circulación materna.
Respecto a EGFR existen trabajos anteriores relacionando un déficit en particulares situaciones como eclampsia, madres fumadoras, etc.30-32 En el presente estudio se ha encontrado una expresión similar en el grupo control de recién nacidos normales y el grupo de RCIU.
El presente estudio es parte integrante de la colaboración de un amplio grupo de investigadores y colaboradores para no sólo analizar causas génicas de la grave situación que para la sociedad significa el nacimiento de un niño con retraso de crecimiento intrauterino, sino que se intenta incidir en las gestantes para que con un seguimiento adecuado del embarazo, una nutrición correcta y eliminación de sustancias deletéreas como alcohol y tabaco colaboren a que el niño siga una curva de crecimiento intrauterino óptima. Está establecida la relación estrecha que existe entre el síndrome X del adulto con complicaciones metabólicas: diabetes, obesidad, hipotiroidismo y un claro retraso intelectual con los antecedentes personales de un retraso de crecimiento intrauterino.